CIENCIAS AGROPECUARIAS-Artículo de Revisión
ARN DE INTERFERENCIA (ARNi): UNA TECNOLOGÍA NOVEDOSA CON POTENCIAL PARA EL CONTROL DE INSECTOS PLAGA
RNA INTEREFERENCE (RNAi): A NOVEL TECHNOLOGY WITH POTENTIAL FOR PEST INSECT MANAGEMENT
Daniel Noriega1, Arnubio Valencia2, Bernardo Villegas3
1 Biólogo, Programa de Biología. Universidad de Caldas, Manizales Caldas, e-mail: daniel.1711114693@ucaldas.edu.co, Calle 65 No 26 - 10, Manizales, Caldas
2Ph.D, Profesor titular, Facultad de Ciencias Agropecuarias. Universidad de Caldas, Manizales, Caldas, e-mail: arnubio.valencia@ucaldas.edu.co Calle 65 No 26 - 10, Manizales, Caldas
3 M.S., Profesor asociado, Facultad de Ciencias Agropecuarias. Universidad de Caldas, Manizales, Caldas, e-mail: bernardo.villegas@ucaldas.edu.coCalle 65 No 26 - 10, Manizales, Caldas
Rev. U.D.C.A. Act & Div. Cient. 19(1): 25-35, Enero-Junio, 2016
RESUMEN
El ARN de interferencia (ARNi) es un mecanismo biológico, ampliamente distribuido en eucariotas, por el cual, se consigue silenciar genes, mediante moléculas de ARN de doble cadena (ARNdc). El descubrimiento de este mecanismo, se llevó hace poco más de 15 años y, desde entonces, se han realizado diferentes investigaciones enfocadas, principalmente, a comprender mejor cómo funciona, su función en diferentes organismos, su uso para describir funciones de genes específicos y las potenciales aplicaciones que tendría en el desarrollo tecnológico, en otras áreas de la ciencia. El silenciamiento de genes se da por la interacción de complejos enzimáticos en el citoplasma con pequeñas moléculas de ARN (siRNA), las cuales, actúan sobre el ARN mensajero (ARNm) endógeno, impidiendo que sea traducido a proteína. Es un hecho que el ARNi puede ser una tecnología alternativa en el control de plagas de importancia agrícola, a través del silenciamiento selectivo de genes, considerados esenciales para la sobrevivencia de la plaga. La implementación de esta tecnología involucra una serie de estudios, que van desde la identificación de genes blanco, el diseño de secuencias de ARNdc, el desarrollo de bioensayos y pruebas en campo, que permitan evidenciar los reales efectos del silenciamiento y la evaluación de factores asociados, que pudieran generar variabilidad del proceso de silenciamiento, investigaciones que son necesarias para que esta tecnología se establezca, finalmente, en el mercado. El presente artículo presenta las bases teóricas del ARNi, los logros de esta tecnología, así como su potencial para el control de insectos plaga.
Palabras clave: Silenciamiento génico, control de plagas, transcriptoma, ARNm.
SUMMARY
RNA Interference (RNAi) is a biological mechanism widely distributed in eukaryotes that allows silencing of genes in the presence of double-stranded RNA (dsRNA) molecules. This mechanism was discovered more than 15 years ago, since then a lot of research projects have been performed in order to obtain a better understanding of how this mechanism works, its function in different organisms, utility for specific gene function description and its potential in the technologic development of other areas. Gene silencing occurs by the interaction between enzymatic complexes in the cytoplasm, with small interference RNA molecules (siRNA), which act on the endogenous messenger RNA (mRNA), therefore preventing their translation to proteins. It is a fact that RNAi could be used as an alternative technology for pest control, through silencing essential genes for the target species. The implementation of this technology involve many necessary studies, ranging from the identification of target genes including dsRNA sequence design, development of bioassays and field tests that show the real effects of dsRNA on gene silencing, and the evaluation of associated factors that could produce variability during the process. Due to this, is necessary more research studies for a marketing establishing of this technology. This paper presents the theory behind the RNAi mechanism, current achievements of this technology, as well as the potential of this technology for insect pest control.
Key words: Gene silencing, pest control, RNA interference, mRNA, transcriptome.
INTRODUCCIÓN
Uno de los avances científicos más relevantes que se han dado en el campo de la biología y la genética molecular, en los últimos 20 años, lo constituye el descubrimiento de un fenómeno biológico, por el cual, se puede interrumpir la expresión de genes específicos, a nivel celular, mediante la introducción de ARN de doble cadena exógeno, un proceso que se denomina silenciamiento génico (Baum & Roberts, 2014). Este mecanismo biológico está siendo considerado prometedor para el control de plagas agrícolas y con potencial en otras áreas de las ciencias aplicadas. La aplicación práctica en laboratorio de este mecanismo tuvo un gran impacto en el estudio de genomas, dado que se logró descubrir la función de muchos genes en diferentes organismos, entre ellos, los insectos (Bellés, 2010), particularmente, en el estudio de la genómica funcional en Drosophila melanogaster (Kuttenkeuler & Boutros, 2004; Chen et al. 2007).
Uno de los primeros indicios de la existencia de este mecanismo endógeno de supresión génica fue encontrado por Napoli et al. (1990), al descubrir una inusual supresión de genes asociados a la coloración en petunias modificadas genéticamente, atribuyendo dicha supresión al gen transgénico de Chalcona sintasa, que ellos modificaron. Más adelante, Guo & Kemphues (1995) describieron un fenómeno similar en Caenorhabditis elegans, al encontrar que individuos tratados de forma independiente con hebras de ARN antisentido y ARN, inhibían la expresión de el gen par-1, lo que resultaba paradójico, ya que se esperaba que aquellos individuos tratados con la hebra sentido, se expresaran normalmente.
Años más tarde, los experimentos de Fire et al. (1998) con C. elegans permitieron elucidar, más detalladamente, la forma como ocurre el proceso de silenciamiento, también conocido como ARN de interferencia (ARNi). Estos investigadores encontraron que el mecanismo por el cual funciona este proceso, se fundamenta en la degradación del ARN mensajero (ARNm) endógeno, a nivel celular, un evento que sucede posterior a la aplicación exógena de ARN de doble cadena (ARNdc), complementario a la secuencia blanco, por lo que el ARNm específico es sujeto a silenciamiento. El mecanismo de silenciamiento de genes fue reportado en años posteriores en un gran número de plantas, de hongos y de animales de diferentes phyla, corroborando la hipótesis de que el mecanismo de ARNi se hallaba ampliamente distribuido en organismos eucariotas (Zou et al. 2003; Dykxhoorn & Lieberman, 2005).
Se han descrito al menos tres funciones principales del mecanismo de ARNi en los organismos eucariotas: la regulación génica específica, la protección contra agentes virales y la defensa del organismo frente a elementos génicos transponibles o trasposones (Sen & Blau, 2006; Billmyre et al. 2013; Liao & Tang, 2015); sin embargo, estas no son las únicas funciones asociadas al ARNi. En plantas, por ejemplo, se han estudiado una variedad de procesos reguladores intracelulares de señalización que ocurren durante el desarrollo fisiológico (Sarkies & Miska, 2014). En mamíferos, por otro lado, se ha reportado una función reparadora del ADN y un sistema de defensa endógeno contra el cáncer (Kole et al. 2012; Ozcan et al. 2015). Estos hallazgos incentivaron nuevos estudios acerca del mecanismo de ARNi y su potencial aplicación en campos, como la agricultura, la medicina y la industria farmacéutica, entre otros (Nandety et al. 2015).
En esta revisión, se presentan los fundamentos y los mecanismos de acción del ARN de interferencia, así como también los aspectos clave de su uso en el manejo de insectos plaga. Se realizó una búsqueda sistematizada de literatura, utilizando sitios web, como Google Scholar, Science Direct y Jstor, con el uso de palabras clave, como RNAi, Gene silencing y pest control, además de la lectura de los artículos más relevantes, citados en previas revisiones. Se analizó la literatura online únicamente en revistas científicas, excluyendo, de esta forma, trabajos de grado o tesis doctorales. Se destacan revistas de temática no especializada, como PLOS One, Science y Nature; revistas especializadas, como Insect Biochemistry and Molecular Biology, Journal of Insect Physiology y Nature Biotechnology, así como también artículos de revisión publicados en revistas, como Nature Reviews y Proceedings of the National Academy of Sciences.
FUNDAMENTOS DEL PROCESO DE ARNi
Pequeños ARN de interferencia (siRNA, por sus siglas en idioma inglés): Fire et al. (1998) encontraron que el uso de ARNdc producía un silenciamiento que perduraba en la siguiente generación, sumado al hecho que solo unas cuantas moléculas eran suficientes para lograr una supresión completa del gen, lo que los llevó a proponer la existencia de un componente amplificador en el proceso de ARNi. Fue entonces cuando se descubrieron los pequeños ARN de interferencia (siRNA), un tipo de ARN no codificante (ARN-nc), constituido por fragmentos de ARNdc, de entre 21 y 25 nucleótidos, los cuales, son producidos a partir del ARNdc, que es suministrado a los individuos tratados (Zamore et al. 2000; Elbashir et al. 2001). El proceso descubierto consiste en la degradación o corte del ARNdc suministrado, hasta convertirse en siRNA, que hibridan en las regiones complementarias del ARNm, evitando que codifique para proteínas específicas (Mello & Conte, 2004). Esto se puede dar de distintas formas, las cuales, serán explicadas más adelante.
Maquinaria enzimática: Una vez se descubrió que el proceso de silenciamiento no se daba de forma directa por la aplicación del ARNdc, se consideró la existencia de una maquinaria intermediaria, que se encargara del proceso de degra dación del ARNdc en siRNA y de la unión de estos últimos fragmentos al ARNm endógeno, para su posterior degradación. Las enzimas implicadas en este mecanismo, se descubrirían poco después por Bernstein et al. (2001), quienes designaron el término ''complejo de silenciamiento inducido por ARN'' (RISC, por sus siglas en idioma inglés), término que se refiere al complejo enzimático, que se encarga de degradar el ARNm.
Una vez el ARNdc entra en la célula, la enzima Dicer, de la familia de las ARNasas tipo III, es la encargada de romper el ARNdc en pequeños fragmentos (siRNA). Además de Dicer, también se han encontrado otros componentes importantes en la maquinaria del ARNi (Dykxhoorn & Lieberman, 2005). Los trabajos llevados a cabo por Martínez et al. (2002) permitieron descubrir la participación de las proteínas argonautas 1 y 2 (Ago1 y Ago2) que, posteriormente, se denominarían Slicer, por su actividad como endonucleasas. Estas proteínas, junto con otras enzimas, forman el complejo RISC y al unirse al ARNm lo degradan mediante motivos catalíticos de ARNasa tipo II, un proceso en el que los siRNAs actúan como guía para que el complejo RISC actúe sobre el ARNm (Bernstein et al. 2003; Whangbo & Hunter, 2008).
Si bien estos son los componentes enzimáticos mejor estudiados, existen otras enzimas que participan en el proceso de silenciamiento. Una de ellas es la polimerasa dependiente de RNA (RdRP, por sus siglas en idioma inglés), la cual, amplifica el silenciamiento para generar un efecto sistémico; sin embargo, en insectos no se ha demostrado, hasta el momento, la presencia de dicha enzima, que tiene un efecto negativo para el control mediante ARNi, ya que únicamente actuaría en las células a las que el ARNdc sea suministrado (Price & Gatehouse, 2008); la importancia de este aspecto radica en los mecanismos de entrada del ARNdc en las células del insecto y la capacidad de generar una respuesta sistémica en el mismo. Según lo propuesto por Whangbo & Hunter (2008) existen dos mecanismos por los que el ARNdc es absorbido en los eucariotas. El primero consiste en el mecanismo de cada célula para generar silenciamiento, que es conocido como ARNi de autonomía celular, ya que se produce tan solo en la célula donde se inyecta o produce el ARNdc. El segundo mecanismo puede actuar en células y en tejidos diferentes a los que produzcan el ARNdc, un mecanismo que ha sido denominado ARNi de autonomía no celular.
ARNi de autonomía celular: Se han descrito dos mecanismos, mediante los cuales, se produce ARNi dentro de las células: uno es el silenciamiento génico post-transcripcional (PTGS, por sus siglas en idioma inglés), que actúa directamente sobre el ARNm de dos formas distintas, generando represión de la traducción, mediante siRNAs, que generan degradación del ARNm en el citoplasma y tienen complementariedad perfecta con el ARNm homólogo o degradando el ARNm, a través de microARNs (miARNs), otro tipo de ARN no codificante, que genera represión de la traducción por medio de complementariedad imperfecta con el ARNm (Castel & Martienssen, 2013; Sarkies & Miska, 2014).
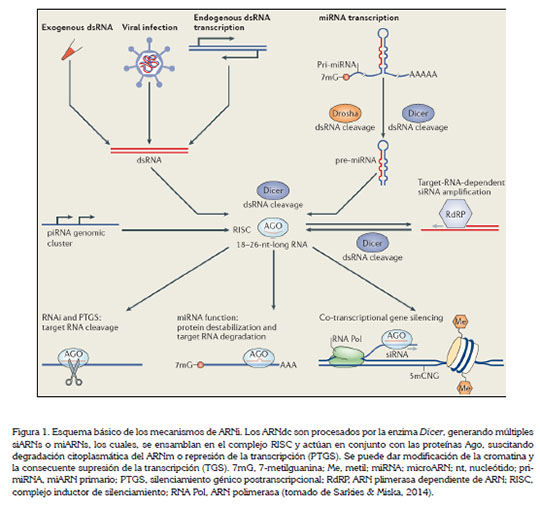
En el proceso mediado por siRNAs, la enzima Dicer rompe selectivamente las largas moléculas de ARNdc, transformándolas en siRNAs (Hammond, 2005; Castel & Martienssen, 2013). Estos siRNAs son incorporados en el complejo RISC, donde la cadena antisentido del siRNA guía de forma específica a la enzima, para producir la ruptura del ARNm homólogo en la porción complementaria, como se puede observar en la figura 1 (Sarkies & Miska, 2014). La ruptura del ARNm se da gracias a las proteínas Argonautas que actúan en conjunto con el complejo RISC, de la siguiente manera: El dominio PAZ (Piwi, Argonauta, Zille) reconoce el extremo terminal con los nucleótidos libres de los siRNAs, mientras que un tercer tipo de ARNnc, denominado PIWI, reconoce y corta en los puntos donde hibridan el siRNA y su ARNm complementario (Lingel et al. 2004; Wang et al. 2009). Cuando el proceso es mediado por miARNs, estos hibridan con el ARNm formando estructuras en bucle que lo desestabilizan, llevándolo, de manera directa, a la degradación citoplasmática (Sarkies & Miska, 2014).

El otro mecanismo de ARNi de autonomía celular, se sabe actúa a nivel del ADN y la cromatina y se conoce como silenciamiento génico dependiente de cromatina (CDGS, por sus siglas en idioma inglés) o silenciamiento génico transcripcional (TGS, por sus siglas en idioma inglés). Este mecanismo se ha estudiado, principalmente, en plantas y en hongos (Guang et al. 2010; Blevins et al. 2014; Holoch & Moazed, 2015), aunque también se ha reportado que ocurre en C. elegans y D. melanogaster (Brennecke et al. 2008; Li et al. 2009; Malone et al. 2009; Burton et al. 2011; Sarkies & Miska, 2014). Consiste en la represión de genes, a nivel transcripcional, por medio de modificaciones epigenéticas en la cromatina, reduciendo la transcripción, mediante la formación guiada de heterocromatina hacia loci específicos. La diferencia fundamental entre este mecanismo y el de PTGS es que en el TGS el complejo efector, conocido como complejo de silenciamiento transcripcional inducido por ARN (RITSC, por sus siglas en idioma inglés), por medio de la unión con factores, que varían dependiendo del organismo, genera modificaciones en las histonas y metilación en el ADN, en los loci, en los cuales, se produce el silenciamiento (Holoch & Moazed, 2015).
ARNi de autonomía no celular: Este mecanismo de absorción del ARNdc puede ser dividido en dos: ARNi ambiental y ARNi sistémico. El primero es aquel proceso, en el cual, una célula es capaz de obtener el ARNdc de su ambiente, mientras que el segundo, involucra un complejo sistema de señalización, a través del silenciamiento, que puede pasar de una célula a otra de forma controlada (Huvenne & Smagghe, 2010). Si bien se ha demostrado en plantas y en C. elegans la existencia de genes que codifican para proteínas encargadas de ejecutar estas funciones de señalización, como es el caso del gen sid-1 que codifica para una proteína transmembranal, en insectos no se ha logrado identificar un sistema de proteínas homólogo que cumpla este papel para la producción de un ARNi sistémico. Por esta razón, las investigaciones para probar ARNi en el control de insectos se han centrado en el intestino, dado que por su alta capacidad de absorción es común observar ARNi ambiental, mediado por endocitosis (Price & Gatehouse, 2008).
Baum & Roberts (2014) mencionan que para lograr una respuesta por ARNi ambiental debe ser completamente operacional y que, hasta el momento, la ruta más efectiva es mediante la ingestión de las secuencias de silenciamiento; sin embargo, algunos órdenes de insectos presentan mecanismos de ARNi sistémico que, hasta ahora, no han sido descritos con tanto detalle, como en C. elegans. Casos, como el de Tribolium castaneu, Apis mellifera y Aphis gossypii, representan algunas especies de insectos, comprobándose la existencia de una señal amplificadora del silenciamiento. Conocer el sistema, mediante el cual funciona dicha señal, permitiría encontrar rutas alternativas a la ingesta directa (Huvenne & Smagghe, 2010). Es importante tener en cuenta estos aspectos y avanzar en investigaciones que ayuden a entender el sistema de amplificación de la señal de silenciamiento, principalmente, para optimizar la entrada y propagación del ARNi en insectos plaga.
POTENCIAL DEL ARNi EN EL CONTROL DE INSECTOS PLAGA
Debido a su alta especificidad, el mecanismo de ARNi tiene un fuerte potencial para uso biotecnológico, considerándose, en los últimos años, como una estrategia para el control de plagas y de enfermedades específicas en la agricultura (Borovsky, 2005; Gordon & Waterhouse, 2007; Whyard et al. 2009; Huvenne & Smagghe, 2010; Wang et al. 2013). La alta demanda de alimentos generada por el constante aumento poblacional en el planeta, sumado a la reducción en el rendimiento y la calidad de los cultivos, ha hecho que la tecnología de ARNi se constituya en una herramienta novedosa para el control de plagas, a nivel mundial y como una opción viable, para superar los obstáculos que existen, para la implementación de las técnicas de transformación molecular convencionales (Yogindran & Rajam, 2015). El mecanismo de ARNi, se presenta como una gran alternativa para el control de insectos plaga, debido, en gran parte, a su especificidad, una de las ventajas de dicha tecnología, para reducir efectos en organismos no blanco y también como un potencial reemplazo a los métodos basados en insecticidas, los cuales, en la mayoría de casos, generan resistencia (Baum & Roberts, 2014).
El interés en la tecnología de ARNi para controlar insectos, se hizo notorio con los trabajos de Baum et al. (2007) (Figura 2A) y Mao et al. (2007), quienes lograron producir plantas transgénicas con resistencia a dos plagas de importancia mundial: el gusano del algodón, Helicoverpa armigera y el gusano de la raíz del maíz, Diabrotica virgifera virgifera. A partir de allí, otros investigadores comenzaron a producir y evaluar secuencias de silenciamiento, dirigidas a genes esenciales para insectos plaga, logrando, en muchos casos, conseguir mortalidad (Zhu et al. 2011; Wang et al. 2013; Xiong et al. 2013; Thakur et al. 2014); sin embargo, como lo resaltan Nandety et al. (2015), existen múltiples parámetros que deben ser estudiados cuidadosamente con anterioridad a su implementación, a nivel agrícola, de forma que se pueda desarrollar una tecnología efectiva, basada en el ARNi. Entre los parámetros más importantes, se tienen: el tipo de inductores de ARNi (siRNAs, miARNs o ARNdc), la concentración más efectiva de dichos inductores y el modo de suministro o liberación del inductor en el organismo plaga. Scott et al. (2013), también resaltan la importancia de los parámetros mencionados y plantea que para el control de insectos plaga, mediante ARNi, también se requiere tener en cuenta, por ejemplo, el tamaño de las secuencias de ARNdc, las cuales, han mostrado ser muy eficientes cuando el tamaño se encuentra en un rango de 50-200 pb; asimismo, plantean que es importante que las secuencias diseñadas tengan la mayor identidad posible con el blanco de ARNm en el insecto. La selección de genes blanco adecuados es otro aspecto que los autores resaltan, evaluando la durabilidad de la proteína codificada y que el transcripto se presente abundantemente.

Selección del blanco para ARNi: En una revisión previa de Nandety et al. (2015), se menciona que los mejores blancos para ARNi son aquellos genes que son parte fundamental en el desarrollo del insecto; por ejemplo, Pitino et al. (2011) y Zha et al. (2011), trabajando con hemípteros plaga, consiguieron reducir la expresión simultánea de varios genes presentes en glándulas salivares y el intestino, respectivamente, logrando la supresión de dichos genes, en un porcentaje mayor al 50%. Xiong et al. (2013), se centraron en la evaluación de secuencias de ARNdc dirigidas, específicamente, a genes involucrados con el metabolismo de la muda en H. armigera, consiguiendo obtener resistencia de la planta al ataque del insecto, pues las larvas que se alimentaron de la planta transformada presentaron tasas de mortalidad, entre el 30 y el 50%, así como también deformaciones en el desarrollo y en la disminución en la tasa de crecimiento de los individuos sobrevivientes.
Si bien, en la mayoría de los casos, se busca producir una mortalidad significativa en el insecto plaga, trabajos como el de Khajuria et al. (2015), demuestran que también es útil escoger genes relacionados con el desarrollo que hagan inviables al insecto. En dicho trabajo, los autores silenciaron en D. virgifera virgifera el gen hunchback (hb), el cual, codifica para un factor de transcripción importante en el desarrollo del plan corporal y también el gen brahma, relacionado con la embriogénesis, logrando producir ausencia de desarrollo embrionario en la descendencia de los organismos tratados.
Genes relacionados con el metabolismo celular y detoxificación, de igual forma, han sido frecuentemente silenciados; ejemplo de ello es el caso exitoso de Baum et al. (2007), mencionado anteriormente. En dicho estudio los investigadores lograron silenciar varios genes codificantes para subunidades de una ATPasa vacuolar, logrando obtener significativas tasas de mortalidad en sus bioensayos. Mao et al. (2011), por otro lado, generaron una planta de algodón transgénica resistente a H. armigera, silenciando el gen CYP6AE14 codificante para la monoxigenasa P450, relacionado con procesos de detoxificación (Figura 2B). Zhu et al. (2012) silenciaron en Cimex lectularius el gen codificante para una proteína multidominio denominada CPR (NADPHCitocromo P450 Reductasa), que está relacionada con una vía metabólica vital en el desarrollo de resistencia a pesticidas, en dicho insecto. Mediante el silenciamiento de dicho gen lograron obtener una significativa reducción de la resistencia del insecto al insecticida deltametrin.
Sin duda alguna, la selección de un blanco adecuado de silenciamiento constituye uno de los aspectos clave para el uso de ARNi, como herramienta para el control de insectos plagas de importancia agrícola. Igualmente, también es una de las tareas más difíciles a la hora de implementar esta estrategia de control, debido, principalmente, a la falta de información genética para muchas de las plagas de interés agronómico (Scott et al. 2013). En este sentido, las herramientas bioinformáticas, se presentan como una gran estrategia para entender, a mayor profundidad, cómo funcionan estos genes en las distintas rutas metabólicas y qué regiones de las secuencias pueden brindar la especificidad necesaria, para luego diseñar, con mayor precisión, secuencias efectivas de silenciamiento; ejemplo de estas herramientas, lo constituye el secuenciamiento de alto rendimiento, como RNA-seq y creación de librerías de ADNc (Koch & Kogel, 2014).
Suministro o liberación de los inductores de ARNi: Una vez escogido un blanco adecuado y diseñado las secuencias para evaluar el ARNi, se debe hacer frente a dos grandes desafíos: encontrar el método efectivo de suministro para los inductores de ARNi y conseguir la estabilidad de los ácidos nucleicos durante o después de la aplicación (Zhang et al. 2010). Lo primero que se requiere es una entrada eficiente de ARNdc o del siRNA, teniendo en cuenta que no en todas las especies de insectos, el silenciamiento, mediante ARNdc, es efectivo. En todo caso, se debe garantizar el suministro de la cantidad mínima necesaria para producir el silenciamiento y que las secuencias de ARNi suministradas no se degraden bajo condiciones de campo o por las enzimas salivares y digestivas, propias del insecto (Baum et al. 2007; Price & Gatehouse, 2008; Liu et al. 2013; Scott et al. 2013).
Un método de suministro de inductores de ARNi ampliamente utilizado ha sido el de la microinyección, a través de la aplicación de secuencias grandes de ARNdc o secuencias cortas de siRNAs, que son sintetizadas in vitro. Casos exitosos de la implementación de esta metodología, para comprobar efectos de mortalidad, interrupción del desarrollo normal o disminución en la alimentación del insecto, se han observado, entre otras especies, en plagas tan importantes, como Manduca sexta, en Levin et al. (2005); Tribolium castaneum, en Tomoyasu et al. (2005); Nilaparvata lugens, en Liu et al. (2010) y Agrilus planipennis, en Zhao et al. (2015). A pesar de que esta forma de suministro es bastante útil en pruebas iniciales de evaluación, porque permite comparar con precisión diferentes concentraciones del inductor y combinar diferentes tipos del mismo, no resulta útil para el control práctico de insectos y está relegada, por el momento, a los estudios básicos y a pruebas en laboratorio (Nandety et al. 2015).
La estrategia más cercana a las condiciones normales de campo, en las que la plaga y el cultivo conviven, es la ingestión directa de las secuencias de silenciamiento. La introducción de ARNdc en dietas artificiales ha sido probada por múltiples autores (Wuriyanghan et al. 2011; Hajeri et al. 2014). Un caso muy exitoso fue el silenciamiento de cinco importantes genes constitutivos en mosca blanca (Bemisia tabaci), logrando obtener altas tasas de mortalidad en el insecto, seis días después de la ingesta de siRNAs y ARNdc. En este trabajo, se comprobó que las secuencias de siRNAs se mantuvieron estables en la dieta del insecto, al menos durante siete días, a temperatura ambiente (Upadhyay et al. 2011). La facilidad con la que se degradan los inductores de ARNi al pasar por el sistema digestivo de los insectos ha impedido que esta estrategia se establezca como una metodología consistente en control de insectos; dado que las dosis empleadas se deben elevar, de tal forma, que se vuelve inviable para uso comercial. En virtud en lo anteriormente planteado, se han propuesto y evaluado nuevas metodologías, para el suministro de las secuencias de silenciamiento (Nandety et al. 2015).
Entre estas nuevas metodologías, se encuentra la liberación de inductores de ARNi, mediada por bacterias y por virus (Koch & Kogel, 2014). Bacterias producidas mediante ingeniería genética son ingeridas por el insecto y, una vez dentro del tracto digestivo, expresan las secuencias de silenciamiento específicas (Nandety et al. 2015). Así, por ejemplo, cepas de Escherichia coli recombinante, expresando secuencias de ARNdc y siRNAs ya se han producido y se han implementado de manera exitosa, como en el caso Zhu et al. (2011), quienes lograron producir mortalidad significativa en Leptinotarsa decemlineata silenciando transcriptos de ARNm con este método.
El silenciamiento mediado por virus, se basa en que el ARNi es el primer mecanismo de defensa de la planta frente a infecciones, de modo que cuando hay una infección en la planta, ésta genera una respuesta sistémica, en la cual, la maquinaria de ARNi de la planta actúa sobre las secuencias de silenciamiento, producidas por el virus. Si dicho virus es modificado genéticamente para que produzca ARNdc específicos contra el insecto plaga, una vez el insecto se alimente de la planta infectada, el virus pasará al tracto digestivo del insecto, donde expresará el ARNdc. Este método es muy prometedor por su eficacia; sin embargo, se debe estudiar con mayor profundidad qué tipo de virus utilizar, dado que muchos de estos virus son potencialmente dañinos para la planta hospedera (Nandety et al. 2015).
A este proceso se le ha dado el nombre de silenciamiento génico inducido por hospederos (HIGS, por sus siglas en idioma inglés) (Koch & Kogel, 2014). El sistema HIGS ha sido utilizado en varios estudios para silenciar genes de importancia metabólica en insectos, obteniendo resultados satisfactorios mediante el empleo de diferentes virus como vectores, principalmente, en plantas de tabaco, en tomate y en cítricos (Khan et al. 2013; Wuriyanghan & Falk, 2013; Hajeri et al. 2014). La estimulación de ARNi en insectos plaga, mediante la generación de plantas transgénicas que expresan inductores específicos, es la estrategia con mejores resultados, en términos de control, en condiciones de campo, tal y como sucede en el caso de especies vegetales, que expresan inductores de ARNi específicos contra el insecto D. virgifera (Baum et al. 2007; Xue et al. 2012).
Otros estudios, en menor proporción, han presentado alternativas diferentes para el suministro de ARNdc. Zhang et al. (2010) utilizaron nanopartículas poliméricas de quitosan ensamblados a secuencias de ARNdc, dirigidas contra genes de quitin sintasa (CHS1 y CHS2), en Anopheles gambiae, las cuales, fueron administradas al insecto, a través del alimento. A pesar de no presentar mortalidad, los individuos tratados aumentaron la susceptibilidad al insecticida diflubenzuron. La aplicación de secuencias de silenciamiento de forma sinérgica con plaguicidas convencionales, con el objetivo que el insecto muestre una mayor susceptibilidad a éstos, constituye una alternativa para conseguir mejores resultados en el control de insectos, empleando menores cantidades del plaguicida, convencionalmente utilizado, y evitando, además de un mayor costo, concentraciones del plaguicida, que resulten nocivas para el mismo cultivo y para el ambiente (Tao et al. 2012; Kim et al. 2015).
Igualmente, el uso de ARNi de forma conjunta con el uso de toxinas tipo Cry de Bacillus thuringiensis (Bt), se podría constituir en una de las estrategias más empleadas en la lucha contra los insectos plaga de cultivos de importancia económica. Es factible suponer que la resistencia que presentan algunas especies de insectos a las toxina tipo Cry de Bacillus thuringiensis (Bt), se podría superar exitosamente, mediante el silenciamiento de los genes que están directamente involucrados en ella, retrasando, de esta forma, la evolución de resistencia al usar diferentes modos de acción (Baum & Roberts, 2014).
CONCLUSIONES Y PERSPECTIVAS FUTURAS
El estudio del mecanismo de ARNi y su posible implementación como tecnología para el control de plagas agrícolas sigue siendo un punto de debate en el medio científico, debido a que aún se presentan muchos vacíos de información para diferentes cultivos e insectos, respecto a las implicaciones en condiciones ambientales y efectividad en largos periodos de tiempo; sin embargo, como se ha visto en esta revisión, numerosos esfuerzos de investigación se han realizado para probar la efectividad de la aplicación de la metodología de ARNi, bajo distintas condiciones, con diferentes organismos y con variados enfoques. Los estudios actuales sobre ARNi han logrado describir una pequeña parte del mecanismo molecular, celular y sistémico, que regula el silenciamiento génico, descubrimientos que han servido para estudiar y conocer la función de cientos de genes de interés. Ese conocimiento, ha permitido manipular la expresión de esos genes en favor del desarrollo de nuevas estrategias de control de plagas y enfermedades de importancia económica mundial; no obstante, se requiere comprender mejor las variaciones del mecanismo entre los diferentes órdenes de insectos y aun entre genes.
Es evidente que el interés en el potencial del ARNi para beneficio del ser humano aumenta con el pasar de los años, conforme continúa el progresivo avance de esta tecnología. Campos de avanzada, como la bioinformática y la biología molecular, se deben considerar las principales aliadas para hacer uso del ARNi, como estrategia exitosa, en el manejo de plagas y de enfermedades de importancia agrícola. La optimización del proceso dependerá de qué tanto avancen las investigaciones en los próximos años, especialmente, en la búsqueda de moléculas de silenciamiento más estables. Asimismo, dependerá de cómo reaccionan las poblaciones de organismos plaga al silenciamiento génico inducido, como también del desarrollo de nuevos métodos de suministro eficiente de las secuencias de silenciamiento.
Agradecimientos: Los autores agradecen a la Vicerrectoría de Investigaciones y Postgrados de la Universidad de Caldas, por el apoyo y la financiación suministrados durante la elaboración del presente artículo. Conflictos de intereses: El manuscrito fue preparado y revisado con la participación de todos los autores, quienes declaramos que no existe conflicto de intereses que ponga en riesgo la validez de los resultados presentados.
BIBLIOGRAFÍA
1. BAUM, J.A.; BOGAERT, T.; CLINTON, W.; HECK, G.R.; FELDMANN, P.; ILAGAN, O.; ROBERTS, J. 2007. Control of coleopteran insect pests through RNA Interference. Nat. Biotechnol. (United Kingdom). 25(11):1322-1326.
2. BAUM, J.A.; ROBERTS, J. 2014. Progress Towards RNAi-Mediated Insect Pest Management. En: Dhadialla, T.; Gill, S. (eds). Insect midgut and insecticidal proteins. Ed. Academic Press (United States). p.249-295.
3. BELLÉS, X. 2010. Beyond Drosophila: RNAi in vivo and functional genomics in insects. Ann. Rev. Entomol. (United States). 55(1):111-128.
4. BERNSTEIN, E.; CAUDY, A.A.; HAMMOND, S.M.; HANNON, G.J. 2001. Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature. (United Kingdom). 409(6818):363-366.
5. BERNSTEIN, E.; KIM, S.Y.; CARMELL, M.A.; MURCHISON, E.P.; ALCORN, H.; LI, M.Z.; HANNON, G.J. 2003. Dicer is essential for mouse development. Nat. Genet. (United Kingdom). 35(3):215-217.
6. BILLMYRE, R.B.; CALO, S.; FERETZAKI, M.; WANG, X.; HEITMAN, J. 2013. RNAi function, diversity, and loss in the fungal kingdom. Chromosome Res. (Netherlands). 21(6-7):561-572.
7. BLEVINS, T.; PONTVIANNE, F.; COCKLIN, R.; PODICHETI, R.; CHANDRASEKHARA, C.; YERNENI, S.; PIKAARD, C.S. 2014. A two-step process for epigenetic inheritance in Arabidopsis. Mol. Cell. (United States). 54(1):30-42.
8. BOROVSKY, D. 2005. Insect peptide hormones and RNA- mediated interference (RNAi): promising technologies for future plant protection. Phytoparasitica. (Netherlands). 33(2):109-112.
9. BRENNECKE, J.; MALONE, C.D.; ARAVIN, A.A.; SACHIDANANDAM, R.; STARK, A.; HANNON, G.J.; 2008. An epigenetic role for maternally inherited piRNAs in transposon silencing. Science. (United States). 322(5906):1387-1392.
10. BURTON, N.O.; BURKHART, K.B.; KENNEDY, S. 2011. Nuclear RNAi maintains heritable gene silencing in Caenorhabditis elegans. Proc. Nal. Acad. Sci. U.S.A. (United States). 108(49):19683-19688.
11. CASTEL, S.E.; MARTIENSSEN, R.A. 2013. RNA interference in the nucleus: roles for small RNAs in transcription, epigenetics and beyond. Nat. Rev. Genet. (United Kingdom). 14(2):100-112.
12. CHEN, M.; DU, Q.; ZHANG, H.; WANG, X.; LIANG, Z. 2007. High-throughput screening using siRNA (RNAi) libraries. Expert. Rev. Mol. Diagn. (United Kingdom). 7(3):281-291.
13. DYKXHOORN, D.M.; LIEBERMAN, J. 2005. The silent revolution: RNA interference as basic biology, research tool, and therapeutic. Ann. Rev. Med. (United States). 56(1):401-423.
14. ELBASHIR, S.M.; LENDECKEL, W.; TUSCHL, T. 2001. RNA interference is mediated by 21-and 22-nucleotide RNAs. Gene. Dev. (United States). 15(2):188-200.
15. FIRE, A.; XU, S.; MONTGOMERY, M.K.; KOSTAS, S.A.; DRIVER, S.E.; MELLO, C.C. 1998. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391(6669):806-811.
16. GORDON, K.H.; WATERHOUSE, P.M. 2007. RNAi for insect-proof plants. Nat. Biotechnol. 25(11):1231-1232.
17. GUANG, S.; BOCHNER, A.F.; BURKHART, K.B.; BURTON, N.; PAVELEC, D.M.; KENNEDY, S. 2010. Small regulatory RNAs inhibit RNA polymerase II during the elongation phase of transcription. Nature. 465(7301):1097-1101.
18. GUO, S.; KEMPHUES, K.J. 1995. par-1, a gene required for establishing polarity in C. elegans embryos, encodes a putative Ser/Thr kinase that is asymmetrically distributed. Cell. (United States). 81(4):611-620.
19. HAJERI, S.; KILLINY, N.; EL-MOHTAR, C.; DAWSON, W.O.; GOWDA, S. 2014. Citrus tristeza virus-based RNAi in citrus plants induces gene silencing in Diaphorina citri, a phloem-sap sucking insect vector of citrus greening disease (Huanglongbing). J. Biotechnol. (Netherlands). 176:42-49.
20. HAMMOND, S.M. 2005. Dicing and slicing: the core machinery of the RNA interference pathway. FEBS Lett. (Netherlands). 579(26):5822-5829.
21. HOLOCH, D.; MOAZED, D. 2015. RNA-mediated epigenetic regulation of gene expression. Nat. Rev. Genet. 16(2):71-84.
22. HUVENNE, H.; SMAGGHE, G. 2010. Mechanisms of dsRNA uptake in insects and potential of RNAi for pest control: a review. J. Insect Physiol. (United Kingdom). 56(3):227-235.
23. KHAJURIA, C.; VÉLEZ, A.; RANGASAMY, M.; WANG, H.; FISHILEVICH, E.; FREY, M.; SIEGFRIED, B. 2015. Parental RNA Interference of Genes Involved in Embryonic Development of the Western Corn Rootworm, Diabrotica virgifera virgifera LeConte. Insect Biochem. Mol. Biol. (United Kingdom). 63(1):54-62.
24. KHAN, A.; ASHFAQ, M.; KISS, Z.; KHAN, A.; MANSOOR, S.; FALK, B. 2013. Use of recombinant tobacco mosaic virus to achieve RNA interference in plants against the citrus mealybug, Planococcus citri (Hemiptera: Pseudococcidae). PLOS One. 8(9):e73657.
25. KIM, Y.H.; ISSA, M.S.; COOPER, A.M.; ZHU, K.Y. 2015. RNA interference: Applications and advances in insect toxicology and insect pest management. Pestic. Biochem. Physiol. (United States). 120:109- 117.
26. KOCH, A.; KOGEL, K.H. 2014. New wind in the sails: improving the agronomic value of crop plants through RNAi‐mediated gene silencing. Plant Bio-technol. J. (United Kingdom). 12(7):821-831.
27. KOLE, R.; KRAINER, A.R.; ALTMAN, S. 2012. RNA therapeutics: beyond RNA interference and antisense oligonucleotides. Nat. Rev. Drug Discovery. (United Kingdom). 11(2):125-140.
28. KUTTENKEULER, D.; BOUTROS, M. 2004. Genomewide RNAi as a route to gene function in Drosophila. Brief. Funct. Gen. Prot. (United Kingdom). 3(2):168-176.
29. LEVIN, D.M.; BREUER, L.N.; ZHUANG, S.; ANDERSON, S.A.; NARDI, J.B.; KANOST, M.R. 2005. A hemocyte-specific integrin required for hemocytic encapsulation in the tobacco hornworm, Manduca sexta. Insect Biochem. Mol. Biol. 35(5):369-380.
30. LI, C.; VAGIN, V.V.; LEE, S.; XU, J.; MA, S.; XI, H.; ZAMORE, P.D. 2009. Collapse of germline piRNAs in the absence of Argonaute3 reveals somatic piRNAs in flies. Cell. 137(3):509-521.
31. LIAO, Y.; TANG, L. 2015. Inducible RNAi system and its application in novel therapeutics. Crit. Rev. Biotechnol. (United Kingdom). 1:1-9.
32. LINGEL, A.; SIMON, B.; IZAURRALDE, E.; SATTLER, M. 2004. Nucleic acid 3'-end recognition by the Argonaute2 PAZ domain. Nat. Struct. Mol. Biol. (United Kingdom). 11(6):576-577.
33. LIU, J.; SMAGGHE, G.; SWEVERS, L. 2013. Transcriptional response of BmToll9-1 and RNAi machinery genes to exogenous dsRNA in the midgut of Bombyx mori. J. Insect Physiol. 59(6):646-654.
34. LIU, S.; DING, Z.; ZHANG, C.; YANG, B.; LIU, Z. 2010. Gene knockdown by intro-thoracic injection of double-stranded RNA in the brown planthopper, Nilaparvata lugens. Insect Biochem. Mol. Biol. 40(9):666-671.
35. MALONE, C.D.; BRENNECKE, J.; DUS, M.; STARK, A.; MCCOMBIE, W.R.; SACHIDANANDAM, R.; HANNON, G.J. 2009. Specialized piRNA pathways act in germline and somatic tissues of the Drosophila ovary. Cell. 137(3):522-535.
36. MAO, Y.B.; CAI, W.J.; WANG, J.W.; HONG, G.J.; TAO, X.Y.; WANG, L.J.; CHEN, X.Y. 2007. Silencing a cotton bollworm P450 monooxygenase gene by plant-mediated RNAi impairs larval tolerance of gossypol. Nat. Biotechnol. 25(11):1307-1313.
37. MAO, Y.B.; TAO, X.Y.; XUE, X.Y.; WANG, L.J.; CHEN, X.Y. 2011. Cotton plants expressing CYP6AE14 double-stranded RNA show enhanced resistance to bollworms. Transgenic Res. (Netherlands). 20(3):665-673.
38. MARTÍNEZ, J.; PATKANIOWSKA, A.; URLAUB, H.; LÜHRMANN, R.; TUSCHL, T. 2002. Single-stranded antisense siRNAs guide target RNA cleavage in RNAi. Cell. 110(5):563-574.
39. MELLO, C.; CONTE, D. 2004. Revealign the world of RNA interference. Nature. 431(7006):338-342.
40. NANDETY, R.S.; KUO, Y.W.; NOURI, S.; FALK, B.W. 2015. Emerging strategies for RNA interference (RNAi) applications in insects. Bioengineered. (United States). 6(1):8-19.
41. NAPOLI, C.; LEMIEUX, C.; JORGENSEN, R. 1990. Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans. Plant Cell. (United States). 2(4):279-289.
42. OZCAN, G.; OZPOLAT, B.; COLEMAN, R.; SOOD, A.; LOPEZ-BERESTEIN, G. 2015. Preclinical and clinical development of siRNA-based therapeutics. Adv. Drug Delivery Rev. (Netherlands). 87:108-119.
43. PITINO, M.; COLEMAN, A.D.; MAFFEI, M.E.; RIDOUT, C.J.; HOGENHOUT, S.A. 2011. Silencing of aphid genes by dsRNA feeding from plants. PLOS One. 6(10):e25709.
44. PRICE, D.R.; GATEHOUSE, J.A. 2008. RNAi-mediated crop protection against insects. Trends Biotechnol. (United Kingdom). 26(7):393-400.
45. SARKIES, P.; MISKA, E.A. 2014. Small RNAs break out: the molecular cell biology of mobile small RNAs. Nat. Rev. Mol. Cell Biol. (United Kingdom). 15(8):525-535.
46. SCOTT, J.G.; MICHEL, K.; BARTHOLOMAY, L.C.; SIEGFRIED, B.D.; HUNTER, W.B.; SMAGGHE, G.; DOUGLAS, A.E. 2013. Towards the elements of successful insect RNAi. J. Insect Physiol. 59(12):1212-1221.
47. SEN, G.L.; BLAU, H.M. 2006. A brief history of RNAi: the silence of the genes. FASEB. J. (United States). 20(9):1293-1299.
48. TAO, X.Y.; XUE, X.Y.; HUANG, Y.P.; CHEN, X.Y.; MAO, Y.B. 2012. Gossypol‐enhanced P450 gene pool contributes to cotton bollworm tolerance to a pyrethroid insecticide. Mol. Ecol. (United Kingdom). 21(17):4371-4385.
49. THAKUR, N.; UPADHYAY, S.K.; VERMA, P.C.; CHANDRASHEKAR, K.; TULI, R.; SINGH, P.K. 2014. Enhanced whitefly resistance in transgenic tobacco plants expressing double stranded RNA of v-ATPase a gene. PLOS One. 9(3):e87235.
50. TOMOYASU, Y.; WHEELER, S.R.; DENELL, R.E. 2005. Ultrabithorax is required for membranous wing identity in the beetle Tribolium castaneum. Nature. 433(7026):643-647.
51. UPADHYAY, S.K.; CHANDRASHEKAR, K.; THAKUR, N.; VERMA, P.C.; BORGIO, J.F.; SINGH, P.K.; TULI, R. 2011. RNA interference for the control of whiteflies (Bemisia tabaci) by oral route. J. Biosc. (India). 36(1):153-161.
52. WANG, Y.; JURANEK, S.; LI, H.; SHENG, G.; WARDLE, G.S.; TUSCHL, T.; PATEL, D.J. 2009. Nucleation, propagation and cleavage of target RNAs in Ago silencing complexes. Nature. 461(7265):754-761.
53. WANG, Z.; DONG, Y.; DESNEUX, N.; NIU, C. 2013. RNAi Silencing of the HaHMG-CoA Reductase Gene Inhibits Oviposition in the Helicoverpa armigera Cotton Bollworm. PLOS One. 8(7):e67732.
54. WHANGBO, J.S.; HUNTER, C.P. 2008. Environmental RNA interference. Trends. Genet. (United Kingdom). 24(6):297-305.
55. WHYARD, S.; SINGH, A.D.; WONG, S. 2009. Ingested double-stranded RNAs can act as species-specific insecticides. Insect Biochem. Mol. Biol. 39(11):824-832.
56. WURIYANGHAN, H.; FALK, B. 2013. RNA Interference towards the potato psyllid, Bactericera cockerelli, is induced in plants infected with recombinant tobacco mosaic virus (TMV). PLOS One. 8(6):e66050.
57. WURIYANGHAN, H.; ROSA, C.; FALK, B.W. 2011. Oral delivery of double-stranded RNAs and siRNAs induces RNAi effects in the potato/tomato psyllid, Bactericerca cockerelli. PLOS One. 6(11): e27736.
58. XIONG, Y.; ZENG, H.; ZHANG, Y.; XU, D.; QIU, D. 2013. Silencing the HaHR3 gene by transgenic plantmediated RNAi to disrupt Helicoverpa armigera development. Int. J. Biol. Sci. (Australia). 9(4):370.
59. XUE, X.Y.; MAO, Y.B.; TAO, X.Y.; HUANG, Y.P.; CHEN, X.Y. 2012. New approaches to agricultural insect pest control based on RNA interference. En: Jockusch, E. (ed). Small RNAs: their diversity, roles and practical uses. Ed. Academic Press (United States). p.73-106.
60. YOGINDRAN, S.; RAJAM, M.V. 2015. RNAi for Crop Improvement. En: Bahadur, B.; Rajam, M.V.; Sahijram, L.; Krishnamurthy, K.V. (eds). Plant Biology and Biotechnology. Ed. Springer India (New Delhi). p.623-637.
61. ZAMORE, P.D.; TUSCHL, T.; SHARP, P.A.; BARTEL, D.P. 2000. RNAi: double-stranded RNA directs the ATPdependent cleavage of mRNA at 21 to 23 nucleotide intervals. Cell. 101(1):25-33.
62. ZHA, W.; PENG, X.; CHEN, R.; DU, B.; ZHU, L.; HE, G. 2011. Knockdown of midgut genes by dsRNA- transgenic plant-mediated RNA interference in the hemipteran insect Nilaparvata lugens. PLOS One. 6(5):e20504.
63. ZHANG, X.; ZHANG, J.; ZHU, K.Y. 2010. Chitosan/double‐stranded RNA nanoparticle‐mediated RNA interference to silence chitin synthase genes through larval feeding in the African malaria mosquito (Anopheles gambiae). Insect. Mol. Biol. (United Kingdom). 19(5):683-693.
64. ZHAO, C.; GONZALES, M.A.A.; POLAND, T.M.; MITTAPALLI, O. 2015. Core RNAi machinery and gene knockdown in the emerald ash borer (Agrilus planipennis). J. Insect Physiol. 72:70-78.
65. ZHU, F.; SAMS, S.; MOURAL, T.; HAYNES, K.F.; POTTER, M.F.; PALLI, S.R. 2012. RNA interference of NADPH-cytochrome P450 reductase results in reduced insecticide resistance in the bed bug, Cimex lectularius. PLOS One. 7(2):e31037-e31037.
66. ZHU, F.; XU, J.; PALLI, R.; FERGUSON, J.; PALLI, S.R. 2011. Ingested RNA interference for managing the populations of the Colorado potato beetle, Leptinotarsa decemlineata. Pest. Manag. Sci. (United Kingdom). 67(2):175-182.
67. ZOU, G.M.; WU, W.; CHEN, J.; ROWLEY, J.D. 2003. Duplexes of 21‐nucleotide RNAs mediate RNA interference in differentiated mouse ES cells. Biol. Cell. (United States). 95(6):365-371.
Recibido: Octubre 14 de 2015 Aceptado: Febrero 4 de 2016
Revista U.D.C.A Actualidad & Divulgación Científica por Universidad de Ciencias Aplicadas y Ambientales se distribuye bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.