CIENCIAS NATURALES-Reporte de Caso
VARIABILIDAD GENÉTICA DE Odocoileus virginianus (Zimmerman, 1970) EN BOYACÁ-COLOMBIA: REPORTE DE CASO
GENETIC VARIABILITY OF Odocoileus virginianus (Zimmerman, 1970) IN BOYACÁ-COLOMBIA: CASE REPORT
Dalia Suárez P.1, Leopoldo Arrieta V.2, Lisette Gaona B.3
1Bióloga. Grupo de Estudios en Genética y Biología Molecular (GEBIMOL). Universidad Pedagógica y Tecnológica de Colombia -UPTC-, Tunja - Boyacá, e-mail: dalia.suarez@uptc.edu.co
2Biólogo M.Sc., Docente, Grupo GEBIMOL, Facultad de Ciencias Básicas, Programa de Biología. Universidad Pedagógica y Tecnológica de Colombia -UPTC-, Tunja - Boyacá, e- mail: leopoldo.arrieta@uptc.edu.co
3Estadística M.Sc., Docente, Grupo GEBIMOL, Facultad de Ciencias Básicas, Programa de Matemáticas y Estadística. Universidad Pedagógica y Tecnológica de Colombia -UPTC-, Tunja - Boyacá, e-mail: lisette.gaona@uptc.edu.co
Rev. U.D.C.A ct. & Di. Cient. 20(2): 485-489, Julio-Diciembre, 2017
https://doi.org/10.31910/rudca.v20.n2.2017.403
INTRODUCCIÓN
Odocoileus viriginianus es una especie con capacidad de adaptabilidad a diferentes ecosistemas, ya que habita desde tierras bajas hasta sistemas montañosos, por encima de los 3.000m de altitud (Ramos et al. 2013). Existen amenazas en la conservación y en el descenso de la densidad poblacional de O. virginianus. La fuerte presión de caza y la transformación drástica de los hábitats ponen en peligro la diversidad genética de sus poblaciones, a nivel nacional (López-Arévalo & González-Hernández, 2006; Martínez Polanco et al. 2015), por tal razón, se pueden originar eventos de selección, tales como: deriva genética, cuello de botella y endogamia a nivel intraespecífico (Hernández et al. 2015). Estos eventos disminuyen la variabilidad genética dentro de las poblaciones, reducen la heterocigosidad y aumentan la probabilidad de transmitir a generaciones futuras, alelos asociados a defectos genéticos. Lo anterior, se traduce en una reducción de la capacidad de adaptación de los animales al medio ambiente y un aumento en el riesgo de extinción (Celis et al. 2010; Ocampo & Cardona, 2013). Para el análisis de la variabilidad genética, a nivel poblacional, se determina la variación existente, intraespecífico o interpoblacional, cuantificando las variaciones o alteraciones en el ADN. Alteraciones que varían desde substituciones de un solo nucleótido hasta mutaciones, que involucran un mayor número de sitios nucleotídicos (Miah et al. 2013). Estas variaciones han sido detectadas implementando el uso de marcadores moleculares, de ADN altamente repetitivo, entre los cuales, se encuentran los microsatélites (SSR). Están formados por secuencias de 1 a 6 pares de bases (pb) nucleotídicas repetidas en tándem y caracterizadas por ser únicas (Deyoung et al. 2009; Miah et al. 2013). En consecuencia, los objetivos de esta investigación fueron: caracterizar los microsatélites Cervid1, Cervid2, Cervid4, Cervid13 y Cervid14 y determinar la variabilidad genética de tres grupos de O. virginianus en semicautiverio, en el departamento de Boyacá, con el fin de obtener información preliminar, para que se establezcan estrategias de conservación de su pool genético, en investigaciones futuras.
MATERIALES Y MÉTODOS
Los casos muestreados fueron: Parque Panaboy, PB, 2 individuos; Colegio Salesiano, CS, 5 individuos y Clínica Biosalud, CB, 3 individuos. Se estudiaron 10 organismos distribuidos de la siguiente forma: cinco machos de diez existentes en CS, un macho y una hembra de dos existentes en PB, dos machos y una hembra de tres existentes en CB. Para la toma de muestra de sangre de los animales, se siguió el protocolo de restricción química y se determinó la dosificación, de acuerdo al peso de los mismos. Se utilizaron dardos (Telinject®) con clorhidrato de ketamina (2 a 4mg/kg) y clorhidrato de xilacina (0,8 a 1,0mg/kg) (Alhuay et al. 2011; Palmer et al. 2017). Se extrajeron 5mL de sangre para la extracción del ADN de las 10 muestras por triplicado, siguiendo el Protocolo del Kit comercial Wizard®-Promega.
La amplificación de los cinco microsatélites, se realizó mediante PCR, en un termociclador (BioRad), a partir de las condiciones tomadas de Dewoody et al. (1995). La Secuencia de los microsatélites y los cebadores usados fueron: Cervid1, (CA)17TA(CA)5: Cebadores F:5'AAATGACAACCCGCTCCAGTATC 3' y R:5'TCCGTGCATCTCAACATGAGTTAG3'; Cervid2,(AC)12AA(AC)7, cebadores F:5'CTTGCTTGGAAAATTCCGTGGAC3', y R:5'TAAACATTGATGGCTTCCCACTCC3'; Cervid4,(CA)17(N)9(AC)2GTT(AC)4, cebadores F:5'ACACCAGAAAGTTCCTCCTCCTG3'y R:5'CGGTCCATAGGGTCACAAAGAGT 3'; Cervid13(CA)100, cebadores F:5'ACAACAGCCACAACAACTGG 3' y R:5' GTTGCCATCCAAATCATTGG3' y Cervid14 (AC)16, cebadores F:5'TCTCTT GCGTCTCCTGCATTGAC3' y R:5'AA TGGCACCCACTCCAGTATTCTTC3'. Los productos, se verificaron en geles de agarosa al 3%, a 110 voltios durante 45 minutos y se fotografiaron en el transiluminador UVP.
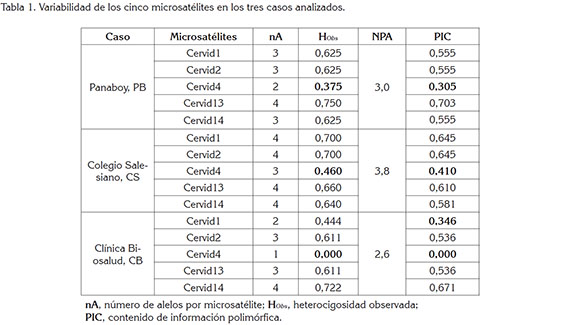
En el procesamiento de datos, para la caracterización de los SSR, se tuvo en cuenta el número de alelos observados por locus (nA), el número promedio de alelos por locus (NPA) y el tamaño de los alelos en pares de bases (pb). En el análisis de variabilidad, se utilizó el programa GenePop versión 1.2 (Raymond & Rousset, 1995) y el programa Molkin versión 3.0 (Gutiérrez et al. 2005), los cuales, permitieron obtener los valores de heterocigosidad observada (Hobs) y el contenido de información polimórfica (PIC).
RESULTADOS Y DISCUSIÓN
Los resultados para la Hobs en el marcador Cervid1, para los casos PB, CS y CB fueron 0,625, 0,700 y 0,444, respectivamente, inferiores a los obtenidos por Deyoung et al. (2003), con 0,830, Miller et al. (2010), con 0,830, Anderson et al. (2002), con 0,840 y Dewoody et al. (1995), con 0,960. Dewoody et al. (1995) reportan en Cervid2 una Hobs, de 0,650 y, para este estudio, los resultados fueron similares, con valores de 0,625, 0,625 y 0,611 en PB, CS y CB, respectivamente. Estos resultados, se pueden atribuir a que algunos de los individuos fundadores de los grupos son heterocigotos: hembra y macho de PB, macho de CS, macho y hembra de CB. El microsatélite Cervid4 está ligado al cromosoma X, lo que genera que los machos analizados sean homocigotos y explicaría la baja Hobs en los grupos PB 0,375, CS 0,460 y CB 0,00. La Hobs en Cervid13 y Cervid14 fue elevada y varió entre 0,611 y 0,750 (Tabla 1).

La variabilidad medida por el NPA fue baja en PB, CS y CB, registrando 3,0, 3,8 y 2,6, respectivamente (Tabla 1). Esta pérdida de diversidad alélica, se puede atribuir a que los grupos fueron conformados con dos o tres individuos provenientes de poblaciones naturales, evidenciando una reducción inicial del número de alelos. Este fenómeno, conocido como efecto fundador, ocasiona endogamia y, por tanto, la pérdida de la variabilidad genética, en las siguientes generaciones. Al ser grupos pequeños, fundados con un número reducido de individuos, el apareamiento entre organismos emparentados es alto, por lo tanto, los valores de endogamia se incrementan. Además, se debe tener en cuenta que en semicautiverio las frecuencias alélicas de las poblaciones se apartan de las condiciones naturales y el flujo genético y la introgresión puede llegar a ser un fenómeno frecuente (Hernández et al. 2015).
El PIC da una medida del grado de información del polimorfismo proporcionado por el microsatélite y la utilidad potencial del mismo, ya que valores inferiores a 0,5 no son útiles para determinar la variabilidad genética de una población (Bejarano et al. 2012). Estos valores, para los cinco marcadores evaluados, en el caso PB, fueron 0,555, para Cervid1, 2 y 14, 0,305, para Cervid4 y 0,703, para Cervid13. Los valores de PIC, en el caso CS, estuvieron entre 0,581, en Cervid14 y 0,645, en Cervid1 y Cervid2, con excepción de Cervid4, que reportó un valor de 0,410.
En el caso CB, los valores de PIC, para los marcadores Cervid2, Cervid13 y Cervid14, estuvieron por encima de 0,500, mientras que Cevid1 y Cervid4 reportaron valores inferiores de 0,346 y 0,000, respectivamente (Tabla 1). Aunque se obtuvieron registros por encima de 0,5 que son informativos, se encuentran por debajo de los hallados por Dewoody et al. (1995), quienes reportaron, en Cervid1: 0,840, Cervid2: 0,650, Cervid4: 0,630 y Cervid14: 0,780. El PIC de Cervid1 es inferior a los valores de Anderson et al. (2002), Deyoung et al. (2003) y Miller et al. (2010), los cuales, reportaron valores de 0,820, 0,830 y 0,980, respectivamente; el PIC bajo en Cervid4, se puede atribuir a que está ligado al cromosoma X. En consecuencia, todos los machos exhiben una sola dosis del microsatélite y se comportan como homocigotos.
En los dos animales analizados, en el caso PB, se observaron 15 alelos en los cinco microsatélites. El marcador Cervid13 presentó cuatro alelos y Cervid4, dos alelos, mientras que Cervid1, Cervid2 y Cervid14 mostraron tres alelos, cada uno. El tamaño de los alelos para Cervid1 osciló entre 155 y 172 pb; para Cervid2, 146 a 221 pb; en Cervid4, de 351 a 361 pb; en Cervid13, de 278 a 353 y en Cervid14, entre 245 a 266.
Para los cinco animales del caso CS, se observaron 19 alelos en los cinco microsatélites. Se encontraron cuatro alelos en cada uno de los microsatélites Cervid1, Cervid2, Cervid13 y Cervid14 y tres alelos, en Cervid4. El tamaño de los alelos, para Cervid1, osciló entre 155 y 187pb; 156 a 244pb, en Cervid2; en Cervid4, de 351 a 361pb; en Cervid13, de 309 a 375pb y en Cervid14, entre 229 y 266pb. En los tres animales evaluados, en el caso CB, se observaron 13 alelos en los cinco microsatélites. El marcador Cervid14 fue el que presentó mayor número de alelos, con cuatro; Cervid2 y Cervid3, tres alelos; Cervid1, dos alelos y un solo alelo, en Cervid4. El tamaño de los alelos para Cervid1 osciló entre 162 y 194pb; 156 a 244pb, en Cervid2; en Cervid4 fue 340pb; en Cervid13, de 309 a 367pb y en Cervid14, entre 234 y 263pb.
El peso del microsatélite Cervid1 en los tres grupos varía entre 155 y 194pb, datos soportados por los valores obtenidos por Anderson et al. (2002), Deyoung et al. (2003) y Miller et al. (2010), quienes reportan valores con rangos similares, 159-193pb, 157-195pb y 187pb, respectivamente. Para los cuatro microsatélites restantes, los pesos obtenidos, únicamente coinciden con los reportados por Leite et al. (2007): Cervid2, 155pb y Cervid4, 356pb, mientras que para Cervid13, 198pb y Cervid14 215pb, son menores.
Con respecto al número de alelos, estos resultados son inferiores a los publicados por Dewoody et al. (1995), quienes reportaron 10 alelos para el microsatélite Cervid1; siete, en Cervid2; cinco, en Cervid4 y ocho, en Cervid14. En las investigaciones de Dewoody et al. (1995), Anderson et al. (2002), Deyoung et al. (2003), Leite et al. (2007), Miller et al. (2010), De la Rosa-Reyna et al. (2012), Lang & Blanchong (2012a fatal prion disease of cervids, was detected in south-central Wisconsin in 2002 and over time has been identified increasingly farther west in the state leading to concerns about CWD spreading to Iowa. Our objective was to characterize genetic connectivity between white-tailed deer (Odocoileus virginianus) y Brommer et al. (2015a fatal prion disease of cervids, was detected in south-central Wisconsin in 2002 and over time has been identified increasingly farther west in the state leading to concerns about CWD spreading to Iowa. Our objective was to characterize genetic connectivity between white-tailed deer (Odocoileus virginianus), los resultados son superiores para Cervid1, en cuanto a nA, que fueron 10, 14, 20, 10, 16, 16, 14 y 7, respectivamente; lo anterior evidencia la pérdida de alelos en los tres casos estudiados. La baja variabilidad alélica encontrada en los grupos PB, CS y CB, se debe al reducido número de individuos y no es producto de la genética de la población natural de donde fueron extraídos, ni tampoco de la hibridación con otros individuos. La pérdida de variabilidad en los grupos no es un resultado sorprendente, dado que cuando una especie se restringe a un área más pequeña y recursos limitados, el tamaño y la diversidad genética se reducen drásticamente (Villanova et al. 2017). Asimismo, los resultados se asocian con lo reportado por Cronin et al. (2006), quienes han dirigido sus investigaciones a determinar los efectos de la domesticación y la cría de fauna en la composición genética de diferentes especies de ciervos. Cronin et al. (2009) consideraron la selección y el paso de las poblaciones de estado natural a estado de cautiverio, como un factor de riesgo, para la pérdida de variabilidad genética, especialmente por fenómenos, como el efecto fundador, la deriva genética, la selección en poblaciones con pequeños tamaños efectivos y el apareamiento no azaroso; sin embargo, algunos de sus resultados han demostrado que la variación genética se puede mantener en poblaciones domésticas y salvajes, con una gestión adecuada, tal como: cruces dirigidos, selección de pie de cría con alta variabilidad y reducción de endogamia.
Se puede concluir que la baja variabilidad de cada uno de los casos analizados demuestra que han pasado por un cuello de botella, producto del establecimiento de grupos en semicautiverio, lo que aumentó el fenómeno de endogamia, por lo tanto, se evidencia una amenaza en la estabilidad genética de los organismos y, por ende, un peligro inminente a su supervivencia. Se recomienda aumentar el pool genético de los grupos, introduciendo individuos con riqueza alélica distinta, provenientes de otras poblaciones y realizar cruces dirigidos entre los individuos, que presentaron el mayor número de alelos en los microsatélites.
Agradecimientos: Agradecemos a la Corporación Autónoma Regional de Boyacá -CORPOBOYACÁ- y a la Universidad Pedagógica y Tecnológica de Colombia -UPTC-, por el apoyo financiero y logístico para el desarrollo de esta investigación. Conflicto de intereses: El manuscrito fue preparado y revisado con la participación de todos los autores, quienes declaramos que no existe conflicto de intereses que ponga en riesgo la validez de los resultados presentados.
BIBLIOGRAFÍA
1. ALHUAY, D.; Li, O.; ALVARADO, A.; FALCÓN, N.; RÍOS, P.; ROJAS, G. 2011. Perfil bioquímico sanguíneo hepático de Venados Cola Blanca (Odocoileus virginianus) criados en cautiverio. Rev. Investig. Vet. Perú. 22(2):138-143.
2. ANDERSON, J.D.; HONEYCUTT, R.L.; GONZALES, R.A.; GEE, K.L.; SKOW, L.C.; GALLAGHER, R.L.; DEYOUNG, R.W. 2002. Development of microsatellite DNA markers for the automated genetic characterization of white tailed deer populations. J. Wildl. Manag. (USA). 66(1):67-74.
3. BEJARANO, D.; PEDRAZA, A.; ROCHA, J.; RODRIGO, M. 2012. Variabilidad genética en subpoblaciones comerciales de la raza criolla colombiana Romosinuano. Corpoica Cienc. Tecnol. Agropec. (Colombia). 13(1):97-107.
4. BROMMER, J.E.; KEKKONEN, J.; WIKSTRÖM, M. 2015. Using heterozygosity-fitness correlations to study inbreeding depression in an isolated population of white-tailed deer founded by few individuals. Ecol. Evol. (USA). 5(2):357-367.
5. CELIS, G.; SÁNCHEZ, N.; LÓPEZ, J. 2010. Evaluación productiva del Venado Cola blanca Odocoileus virginianus (Zimmermann, 1780) en cautiverio. Rev. Fac. Ciencias Agropec. (Colombia). 2(1):19-28.
6. CRONIN, M.A.; MACNEIL, M.D.; PATTON, J.C. 2006. Mitochondrial DNA and microsatellite DNA variation in domestic reindeer (Rangifer tarandus tarandus) and relationships with wild caribou (Rangifer tarandus granti, Rangifer tarandus groenlandicus, and Rangifer tarandus caribou). J. Heredity. (UK). 97:525-530.
7. CRONIN, M.A.; RENECKER, L.A.; PATTON, J.C. 2009. Genetic variation in domestic and wild elk (Cervus elaphus). J. Anim. Sci. (USA). 87:829-834.
8. DE LA ROSA-REYNA, X.; CALDERON-LOBATO, R.D.; PARRA-BRACAMONTE, G.M.; SIFUENTES-RINCON, A.M.; DEYOUNG, R.W.; GARCIA-DE LEON, F.J.; ARELLANO-VERA, W. 2012. Genetic diversity and structure among subspecies of white-tailed deer in Mexico. J. Mammal. (UK). 93(4):1158-1168.
9. DEWOODY, J.A.; HONEYCUTT, R.L.; SKOW, L.C. 1995. Microsatellite Markers in White-Tailed Deer. J. Heredity. 86(4):1994-1996.
10. DEYOUNG, R.W.; DEMARAIS, S.; HONEYCUTT, R.L.; ROONEY, A.P.; GONZALES, R.A.; GEE, K.L.; ANDERSON, J.D. 2003. Evaluation of a DNA microsatellite panel useful for genetic exclusion studies in white-tailed deer. Wildl. Soc. Bull. (USA). 31(1):220-232.
11. DEYOUNG, R.W.; DEMARAIS, S.; GEE, K.L.; HONEYCUTT, R.L.; MICKEY, W.; GONZALES, R.A. 2009. Molecular evaluation of the White-tailed Deer (Odocoileus virginianus). J. Mammal. 90(4):946-953.
12. GUTIÉRREZ, J.P.; ROYO, L.J.; ÁLVAREZ, I.; GOYACHE, F. 2005. MolKin v2.0: a computer program for genetic analysis of populations using molecular coancestry information. J. Hered. 96:718-721.
13. HERNÁNDEZ, P.M.; PARRA, G.M.; DE LA ROSA, X.F.; CHASSIN, O.; SIFUENTES, A.M. 2015. Genetic shifts in the transition from wild to farmed white-tailed deer (Odocoileus virginianus) population. Int. J. Biodivers. Sci. Ecosyst. Serv. Manage. (UK) 10(1):3-8.
14. LANG, K.R.; BLANCHONG, J.A. 2012. Population genetic structure of white-tailed deer: Understanding risk of chronic wasting disease spread. J. Wildl. Manag. 76(4):832-840.
15. LEITE, K.C.; COLLEVATTI, R.G.; MENEGASSO, T.R.; TOMAS, W.M. 2007. Transferability of microsatellite loci from Cervidae species to the endangered Brazilian marsh deer, Blastocerus dichotomus. Genet. Mol. Res. (Brasil). 6(2):325-330.
16. LÓPEZ-ARÉVALO, H.; GONZÁLEZ-HERNÁNDEZ, A. 2006. Venado sabanero Odocoileus virginianus En: Libro rojo de los mamíferos de Colombia. Serie Libros Rojos de especies amenazadas en Colombia 114-120. Conservación internacional y ministerio de ambiente y desarrollo territorial. Bogotá.
17. MARTÍNEZ POLANCO, M.F.; MONTENEGRO, O.L.; PEÑA L., G.A. 2015. La sostenibilidad y el manejo de la caza del venado cola blanca (odocoileus virginianus) por cazadores recolectores del periodo precerámico de la sabana de Bogotá, en el yacimiento arqueológico de Aguazuque (Colombia). Caldasia. 37(1):1-14.
18. MIAH, G.; RAFII, M.Y.; ISMAIL, M.R.; PUTEH, A.B; RAHIM, H.A.; ISLAM, KH.N.; LATIF, M.A. 2013. A review of microsatellite markers and their applications in rice breeding programs to improve blast disease resistance. Int. J. Mol. Sci. (Suiza). 14: 22499-22528.
19. MILLER, B.F.; DEYOUNG, R.W.; CAMPBELL, T.A.; LASETER, B.R.; MARK, W.; MILLER, K.V. 2010. Fine-scale genetic and social structuring in a central Appalachian white-tailed deer herd. J. Mammal. 91(3):681-689.
20. OCAMPO, R.G.; CARDONA, H.C. 2013. La endogamia en la producción animal. Rev. Col. Cienc. Anim. (Colombia). 5(1):465-479.
21. PALMER, M.V.; COX, R.J.; WATERS, W.R.; THACKER, T.C.; WHIPPLE, D.L. 2017. Using white-tailed deer (Odocoileus virginianus) in infectious disease research. J. Am. Assoc. Lab. Anim. Sci. (USA). 54(4):1-11.
22. RAMOS, M.; GALLINA, S.; MANDUJANO, S. 2013. Habitat and human factors associated with white-tailed deer density in the tropical dry forest of TehuacánCuicatlán Biosphere Reserve, Mexico. Trop. Conserv. Sci. (USA). 6(1):70-86.
23. RAYMOND, M.; ROUSSET, F. 1995. GENEPOP (version 1.2): Population genetics software for exact tests and ecumenicism. J. Heredity. (UK). 86:248-249.
24. VILLANOVA, V.L.; HUGHES, P.T.; HOFFMAN, E.A. 2017. Combining genetic structre and demographic analyses to esimate persistence inendangered key deer (Odocoileus virginianus clavium). E.A Conserv. Genet. (Holanda). doi:10.1007/s10592-017-0958-2.
Recibido: Octubre 31 de 2016 Aceptado: Noviembre 23 de 2017

Revista U.D.C.A Actualidad & Divulgación Científica por Universidad de Ciencias Aplicadas y Ambientales se distribuye bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.