CIENCIAS AGROPECUARIAS-Artículo Científico
DESARROLLO DE LA TÉCNICA DE INTERFERÓN GAMMA (IFN-γ) PARA LA DETECCIÓN DE BOVINOS INFECTADOS CON Listeria monocytogenes
TECHNICAL DEVELOPMENT OF GAMMA INTERFERON (IFN-γ) FOR DETECTION OF Listeria monocytogenes INFECTED CATTLE
Carolina Gallego1, Manuel I. Gallego2, José L. Azumendi3, Jeimara Paipa4, Diana Jaramillo5
1 MV, M.Sc, c.Ph.D., Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, Calle 222 No. 55-37, Bogotá, Colombia, e-mail: cgallego@udca.edu.co
2 MV, M.Sc, Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, Calle 222 No. 55-37, Bogotá, Colombia, e-mail: mgallego@udca.edu.co
3 MV, Fundación Colombiana de Estudios de Parásitos, Calle 137 No. 12-15, Bogotá, Colombia, e-mail: azumendij@yahoo.com
4 MV, Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, Calle 222 No. 55-37, Bogotá, Colombia, e-mail: jeimara87@hotmail.com
5 MV, Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, Calle 222 No. 55-37, Bogotá, Colombia, e-mail: nanacarolina@hotmail.com
Rev. U.D.C.A Act. & Div. Cient. 18(1): 163-170, Enero-Junio, 2015
RESUMEN
El objetivo de esta investigación fue estandarizar el protocolo técnico para la cuantificación de IFN-γ de bovinos infectados con L. monocytogenes. El proyecto, se realizó en un conglomerado lechero del departamento de Cundinamarca. Se seleccionaron 169 animales, a los cuales, se les realizó aislamiento microbiológico, a partir de materia fecal, leche e hisopados vaginales, encontrando 53 positivos y 116 negativos; posteriormente, se tomaron muestras de sangre completa con heparina, para cuantificar, por ELISA, el IFN-γ producido después de la sensibilización de linfocitos con listeriolisina O y, finalmente, se realizó una comparación de los resultados obtenidos por aislamiento microbiológico con la cantidad de INF- γ producido. De acuerdo con el análisis estadístico, el promedio de la medición de IFN-γ bovino de los animales negativos por aislamiento microbiológico fue de 12,75, con una varianza de 1199.93 y de los animales positivos por aislamiento microbiológico fue de 1,52, con una varianza de 316,65. El análisis de varianza y la prueba F de Fisher demostraron una relación estadísticamente significativa entre la cantidad de IFN-γ con el aislamiento microbiológico de animales positivos y negativos (P<0,05). De acuerdo con los resultados obtenidos, la técnica de ELISA para IFN-γ permite la detección indirecta de animales infectados con L. monocytogenes y la posibilidad de establecer medidas tempranas de control y de prevención.
Palabras clave: Listeriosis, interferón, diagnóstico, ganado de leche.
SUMMARY
The objective of this research was to standardize the final technical protocol for quantification of IFN-γ in cattle infected with L. monocytogenes. The project was conducted in a dairy conglomerate of Cundinamarca. One hundred sixty nine animals were selected which underwent microbiological isolation from faeces, milk and vaginal swabs finding 116 positive animals and 53 negative, then, samples of whole blood with heparin were obtained to quantify IFN-γ produced after T linfocytes priming with listeriolysin O by ELISA and, finally, the results obtained by microbiological isolation were compared with the amount of IFN-γ produced. According to the statistical analysis the average measuring of INF-γ bovine in negative animals by microbiological isolation was12.75 with a variance of 1199.93 and the average measuring of INF-γ bovine in positive animals was 1.52 with a variance of 316.65. The analysis of variance and Fisher's F-test showed a statistically significant relationship between the amount of IFN-γ with microbiological isolation (P <0.05), the sensitivity of the IFN-γ technique was 73-100% and specificity 85-97.6%. According to the results obtained by ELISA IFN-γ, it allows indirect detection of animals infected with L. monocytogenes and the possibility of early action and prevention control.
Key words: Listeriosis, interferon, diagnosis, dairy cattle.
INTRODUCCIÓN
Listeria monocytogenes es un patógeno de gran importancia en salud pública y en salud animal, debido a su capacidad de sobrevivir en diferentes ambientes y, por lo tanto, hacer de ella una bacteria ubicua, permitiéndole hacer parte de la cadena alimenticia del humano al contaminar carne y productos lácteos e, inclusive, otros alimentos de origen no animal, como los vegetales (Tienungoon et al. 2000; Borucki & Peppin, 2003; Paillard & Dubois, 2003; Czuprynsky, 2004; Moltz & Martin, 2005; Pineda & Mora, 2006; Pérez-Trallero et al. 2014). En los seres humanos induce una serie de síndromes que pueden ir desde la presentación de abortos hasta septicemia y meningitis (Pineda & Mora, 2006), afectando predominantemente mujeres gestantes, pacientes inmunocomprometidos e individuos viejos (Williams et al. 2012).
La enfermedad en los bovinos cursa también con diferentes síndromes que varían de acuerdo con el tropismo bacteriano; se ha reportado la capacidad de la bacteria de migrar hasta el sistema nervioso central e inducir lesiones compatibles con meningoencefalitis de carácter mononuclear, con formación de microabscesos (Czuprynski, 2004; Bubonja et al. 2006); también se ha reportado su capacidad de invadir otros órganos y provocar lesiones, como necrosis multifocal e infiltrado inflamatorio del bazo e hígado (Czuprynski, 2004; Bubonja et al. 2006; Pineda & Mora, 2006). Una de las principales consecuencias de la infección con L. monocytogenes en bovinos es la inducción de abortos e invasión del feto y la placenta, como consecuencia del proceso de septicemia resultante de la infección.
El diagnóstico etiológico de listeriosis en el laboratorio clínico se hace mediante el aislamiento microbiológico, a partir de muestras estériles de sangre, de líquido cefalorraquídeo y amniótico, de placenta y de materia fetal, el cual, es altamente dispendioso y puede arrojar resultados falsos negativos (Sierra et al. 2004; Torres et al. 2005; Vanegas et al. 2006). Otra técnica diagnóstica reportada para la detección de animales con listeriosis es la serología, específicamente, la técnica de ELISA (Baetz et al. 1996; Gasanov et al. 2005; Elezebeth et al. 2007; López-Expósito et al. 2008); sin embargo, se debe tener en cuenta que la resistencia del hospedero contra L. monocytogenes es mediada por inmunidad celular y los anticuerpos podrían indicar infecciones no activas (Pineda & Mora, 2006).
En Colombia, en el 2003, Gallego et al. (2003) encontraron un porcentaje de positividad del 2,4 y el 24% a L. monocytogenes, en un conglomerado lechero de la Sabana de Bogotá y Moreno et al. (2007) reportaron una positividad en leche también por aislamiento microbiológico, del 92,8%, en el departamento de Boyacá. Asimismo, Gallego et al. (2007) consiguieron aislamientos positivos del 2,26% en canales de ganado Holstein y en canales de ganado cebú, del 8,33%, en una planta de beneficio de la Sabana de Bogotá (Villamil et al. 2007).
La listeriolisina (LLO) ha sido considerada el mayor factor de virulencia de L. monocytogenes importante en la invasión celular, la sobrevivencia intracelular y la diseminación célula a célula, además de procesos previos a la entrada de la bacteria (Vera et al. 2013).
Diferentes investigadores han demostrado la capacidad de L. monocytogenes de inducir la producción de interferón-γ endógeno en infecciones tempranas, lo cual, le conferiría resistencia al animal contra la infección (Nakane et al. 1990). El INF-γ o IFN II es secretado por células CD8T, por el subconjunto Th1 de células CD4T y por células natural killer (NK). Dentro de las funciones del IFN-γ están la estimulación de la actividad fagocítica y la regulación de la presentación de antígenos, a través de las moléculas del complejo mayor de histocompatibilidad tipo I y II. La resistencia contra bacterias intracelulares, como L. monocytogenes, es dependiente de IFN-γ, que es fuertemente secretada en las primeras fases de infección (Skoberne, & Geginat, 2002; Ling et al. 2008; Czuprynsky et al. 2010).
Los macrófagos han sido el foco de la inmunidad innata durante la infección con L. monocytogenes, ya que la primera replicación bacteriana ocurre dentro de ellos, quienes, en respuesta a la infección, secretan FNT-α e IL-12; estas dos citoquinas inducen a las células NK a producir IFN-γ, la cual, a su vez, lleva a la activación de los macrófagos para mejorar su capacidad fagocítica (Zenewicz & Shen, 2007).
La técnica de diagnóstico llamada ensayo de IFN-γ ha sido ampliamente empleada para el diagnóstico de patógenos con ciclos de vida intracelulares, como Mycobacterium tuberculosis (Cho, 2007; Hansted et al. 2007; Madariaga et al. 2007) y consiste en la detección de la producción de IFN-γ por parte de los linfocitos provenientes de animales con infecciones recientes y latentes. Diferentes autores han empleado esta técnica para el estudio de la listeriosis en estudios de experimentación in vitro e in vivo, pero no se conocen referencias del empleo de esta técnica en animales con infección natural en campo (Nakane et al. 1990; Geginat et al. 1999; Kim et al. 2001; Skoberne et al. 2001; Skoberne & Geginat, 2002; Kamm et al. 2003).
Las ventajas que el ensayo de IFN-γ ofrece frente a otras técnicas de diagnóstico, se podrían resumir en los siguientes aspectos (Madariaga et al. 2007): Detección indirecta de la presencia del microorganismo, mediante la cuantificación del IFN-γ como indicador de la respuesta inmune celular; detección de animales con infecciones activas, que permitiría detectar, rápidamente, los animales infectados, mejorando el control de la enfermedad, al limitar la comercialización de sus productos e implementar medidas oportunas de control; la muestra para llevar a cabo la técnica es sangre completa con anticoagulante, es fácil de obtener y no implica un manejo agresivo del animal; el procesamiento de la muestra toma aproximadamente 24 horas, lo que garantiza una entrega rápida de resultados y la prontitud en la toma de decisiones; adicionalmente, se pueden analizar muestras de varios animales simultáneamente.
La presente investigación tuvo como objetivo estandarizar el protocolo técnico para la cuantificación de IFN-γ producido de bovinos infectados con L. monocytogenes, para poder detectar rápidamente animales infectados, mejorando el control de la enfermedad, al limitar la comercialización de sus productos e implementar medidas terapéuticas a tiempo.
MATERIALES Y MÉTODOS
El estudio, se llevó a cabo en hatos de un conglomerado lechero situado en un área comprendida por los municipios de Madrid, Mosquera y El Rosal, del departamento de Cundinamarca, en los cuales, en una investigación anterior, se había encontrado una prevalencia del 24%, por aislamiento microbiológico (Gallego et al. 2003).
Con base en una población animal de 25.000 animales (Castro, 2010) y de acuerdo al diseño estadístico, se seleccionó una población de 169 vacas raza Holstein o cruces con esta raza, en producción y capacidad reproductiva, pertenecientes a diferentes fincas lecheras (Kish, 1972; Kuehl, 2001). El tamaño de la muestra fue obtenido considerando poblaciones finitas, con un tamaño de población de 25.000 animales, nivel de confianza, 95%, error aceptado, 5% y prevalencia estimada, 24%.
Se tomaron muestra de leche, materia fecal e hisopados vaginales, del total de animales seleccionados. Estas muestras fueron inmediatamente sembradas en caldo PALCAM® Listeria (Britania, Argentina) y conservadas a 4°C, durante 48 a 72 horas y luego transportadas al laboratorio para continuar con el aislamiento e identificación de los microorganismos; posteriormente, se transfirieron en agar PALCAM® Listeria (Britania, Argentina) e incubaron a 37°C, por 5 a 8 días (Gallego et al. 2003). Para confirmar los aislamientos positivos o sospechosos a L. monocytogenes en medio PALCAM, se realizó un tercer repique de dichos aislamientos en placas 3MTM PetrifilmTM (3M Microbiología, USA), para Listeria (Luca, 2005). Durante el proceso de aislamiento e identificación de los cultivos, se empleó, como control, una cepa de Listeria monocytogenes proveniente de un aislamiento clínico, donado por el Laboratorio de Microbiología de la Universidad Javeriana.
Después de la incubación, se analizó la presencia de colonias típicas de Listeria spp. en los medios de cultivo descritos previamente; se realizó tinción de Gram, para identificar los bacilos Gram positivos pleomórficos, cortos y delgados.
Para finalizar con el proceso de identificación de la bacteria, se aplicó la prueba de Christie-Atkins--Munch-Peterson (CAMP), hemólisis, movilidad a 25°C, fermentación de D-manitol, L-ramnosa y D-xilosa y reducción de nitratos, para confirmar la presencia de Listeria monocytogenes (Luca, 2005; Manual de la OIE, 2004).
Se tomaron muestras de sangre completa con heparina de los bovinos previamente identificados como negativos y positivos a L. monocytogenes, mediante aislamiento microbiológico; una vez tomada la muestra, se llevaron al laboratorio a 4°C, en un período inferior a ocho horas, para la determinación cuantitativa del IFN-γ bovino por la técnica de ELISA (Novex ® Méjico).
Para la extracción de la listeriolisina y titulación de la misma, se siguió la metodología de Domínguez et al. (1986), empleando, como control, una cepa de L. monocytogenes, suministrada por el Laboratorio de Microbiología de la Universidad Javeriana. Inicialmente, se realizó un cultivo de L. monocytogenes de la cepa control, en caldo BHI, a 37°C, durante 24 horas y, mediante un equipo de filtración, se recolectaron 100mL, que fueron pasados por una membrana de 0,45µm, en condiciones de esterilidad. El sobrenadante de la primera filtración, se pasó por un filtro de un tamaño de poro de 0,22µm, en condiciones de esterilidad (Domínguez et al. 1986).
Para realizar la titulación de la listeriolisina O (LLO), se tomó sangre de cordero para la obtención de eritrocitos, que fueron lavados previamente en buffer PBS 1X, pH 7,4 y fueron ajustados a una concentración de 3%.
En 12 pozos de una caja de microtécnica, se adicionaron 50µL de solución salina al 0,85%; en otros 12 pozos, 50µL de caldo BHI estéril y, en otros 12 pozos, 50µL de la solución filtrada que contiene LLO y se adicionaron 50µL de eritrocitos de cordero al 3%, en todos los pozos. Se incubó durante 1 hora, a 37°C y se evaluó la ausencia o presencia de hemólisis por la presencia de la toxina (β-hemólisis).
El titulo de la actividad hemolítica, se expresó como Unidades Hemolíticas Completas (UHC), las cuales, son consideradas como el recíproco de la dilución más alta, a la cual, el 100% de la hemólisis se evidencia y, Unidades Hemolíticas Mínimas (UHM), como el recíproco de la dilución más alta, en la que se detectó la hemólisis (Domínguez et al. 1986). Para lograr la sensibilización de las células linfocitarias de los animales en estudio, se enfrentó 1mL de sangre heparinizada, de cada uno de ellos, con 100µL de listeriolisina O en dilución 1:2; asimismo, se separó 1mL de la misma sangre sin sensibilización y la incubación se realizó en tubos de ensayo durante 18 horas, a 37°C. Pasado el tiempo de incubación, se centrifugaron las muestras a 2200 gravedades, por 10 minutos; el plasma obtenido, se conservó a -20°C, hasta que fueron procesadas de acuerdo a la técnica de ELISA (Domínguez et al. 1986).
La técnica de ELISA para IFN-γ, se efectuó según el siguiente protocolo: las microplacas de ELISA de 96 pozos fueron sensibilizadas por un anticuerpo monoclonal específico para el IFN-γ bovino; este anticuerpo permite la captura específica del IFN-γ bovino, que se encuentra presente en el plasma de los animales problema.
En cada pozo de la microplaca, se adicionaron 100µL de suero control y cantidades similares de suero sin sensibilizar y suero sensibilizado, todos por duplicado; se incubaron por 1 hora y pasado el tiempo, se realizó el lavado 3 veces con solución de lavado, a una concentración 20X, de acuerdo con las instrucciones dadas por la casa matriz. Luego, se preparó la dilución de la solución Buffer, en una concentración 5X y se empleó para preparar el conjugado, que se llevó a una concentración 50X; de esta última solución, se añadieron 100µL a cada pozo y, nuevamente se incubó, durante 1 hora, a temperatura ambiente y se repitieron los lavados, como se describió anteriormente. Por último, se preparó la solución sustrato con el cromógeno y se colocaron 100µL de dicha solución a cada pozo y se incubaron por 10 minutos. Pasado este tiempo, se adicionaron 50µl de solución frenadora a cada pozo y se llevaron inmediatamente al lector de ELISA, con filtro de 450 a 620nm, para realizar la lectura y análisis de los resultados.
La información de animales positivos y negativos al examen microbiológico y positivos y negativos a la prueba de ELISA para IFN-γ fue descrita, a través de metodología univariada, posteriormente, fue verificada la existencia de relación entre resultados (examen microbiológico vs. prueba ELISA IFN-γ), por medio de la prueba Chi cuadrado y, finalmente, se verificó la existencia de similitudes o diferencias en la respuesta, por el análisis de la varianza de una vía (Prueba F), con los software EPIINFO TM (2014) y R (R Core Team, 2014).
RESULTADOS Y DISCUSIÓN
Las infecciones por L. monocytogenes se caracterizan por inducir cuadros reproductivos, nerviosos y sistémicos muy severos, que pueden afectar, tanto a la población humana como animal. Las características microbiológicas y patogénicas de este género bacteriano hacen que su diagnóstico sea dispendioso y los resultados no siempre son satisfactorios. Por lo tanto, la necesidad de desarrollar nuevas herramientas para el diagnóstico de la enfermedad se hace evidente. L. monocytogenes es un microorganismo con capacidad de internalización en las células del hospedero, característica que induce respuestas específicas del sistema inmune mediadas por INF-γ, razón por la cual, en esta investigación, se indagó sobre la producción de INF-γ en bovinos infectados con L. monocytogenes.
En este estudio, se incluyeron 169 animales, de los cuales, 116 resultaron negativos para L. monocytogenes, por aislamiento microbiológico y 53 animales fueron positivos, 31,36%, a partir de muestras de materia fecal y de éstos, el 7,74% (13), lo fueron también a partir de muestras leche, lo que indica una prevalencia del 31,36%. Los resultados de estos estudios podrían confirmar los altos niveles que puede alcanzar la infección por L. monocytogenes en los productos y subproductos de origen animal para los seres humanos y animales y justifican los esfuerzos para la implementación de técnicas, que permitan el diagnóstico de la enfermedad.
En la presente investigación fue empleado el ensayo de IFN-γ (Cho, 2007; Dheda et al. 2007; Hansted et al. 2007; Madariaga et al. 2007), para la detección de animales positivos a L. monocytogenes. Diferentes autores han empleado esta técnica para el estudio de la listeriosis in vitro e in vivo, pero no se conocen referencias del empleo de esta técnica en animales con infección natural en campo (Nakane, et al. 1990; Geginat, et al. 1999; Kim et al. 2001; Skoberne, et al. 2001; Skoberne & Geginat, 2002; Kamm et al. 2003).
Los resultados de la prueba de actividad hemolítica indicaron que la concentración de hemolisina óptima estaba en una dilución 1:2. Los resultados de la calibración y estandarización de la técnica de ELISA para la cuantificación de INF-γ bovino reportan, de acuerdo con los estadísticos de regresión, un R2 de 0,96, para la curva de estandarización de INF-γ bovino puro y un coeficiente de correlación múltiple de 0,98, lo que indica que existe una alta relación entre la respuesta de INF-γ y el análisis microbiológico.
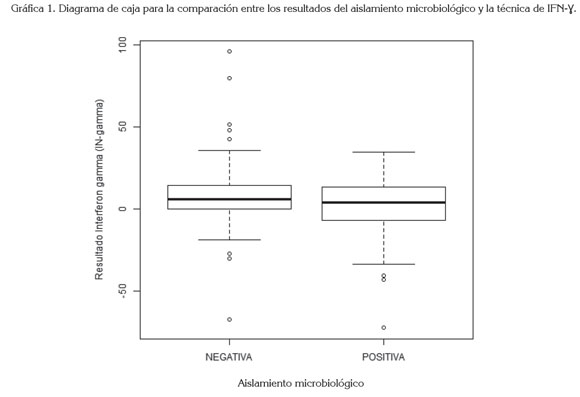
Los datos previamente descritos se analizaron, a través de metodologías de estadística descriptiva, como la representación de la curva de densidad sobre el histograma del comportamiento de la variable, diagramas de caja comparativos para determinar similitudes en la varianza (Grafica 1); se utilizó, también, el estadístico de prueba chi cuadrado, para determinar si existía o no relación entre la respuesta del aislamiento positivo y la medición de INF-γ. Adicionalmente, para someter los datos a metodologías explicativas más robustas, se probaron los supuestos de normalidad de errores y la homogeneidad de las varianzas entre grupos, aceptándose la Ho para el segundo supuesto, razón por la cual, se procede a efectuar el análisis de varianzas (prueba F), en pro de verificar si existe una relación estadísticamente significativa entre los resultados del aislamiento microbiológico y la medición de INF-γ por la técnica de ELISA.

De acuerdo con la estadística descriptiva, se encontró que el promedio de la medición de INF-γ bovino de los animales negativos por aislamiento microbiológico fue de 9,4, con error estándar de 3,46 y de los animales positivos por aislamiento microbiológico fue de 1,4, con error estándar de 2,26 (Tabla 1), donde se aprecia que la respuesta media del aislamiento positivo es ligeramente menor a la del aislamiento negativo. Adicionalmente, se registra que la variación global de la respuesta en el resultado positivo es superior a la del negativo, confirmándose lo reportado en el coeficiente de variación (CV), de la tabla 2, sugiriendo que los datos no son homogéneos en ninguno de los casos (CV > 0,2).
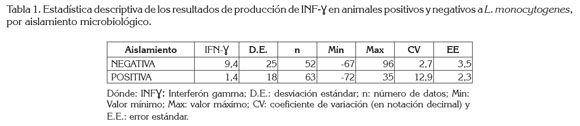

Comparando si existe una relación entre las pruebas aplicadas a los individuos, los resultados obtenidos del aislamiento y la producción de INF-γ, se encuentra mediante la prueba chi cuadrado, una relación altamente significativa entre los dos resultados; esto implica, que efectivamente utilizar una u otra metodología, da como diagnóstico la misma condición (Tabla 1).
Finalmente, a través de la prueba F (Análisis de la varianza de una vía), se halla que la media de resultados positivos y negativos de IFN-γ es la misma (p<0.05153) (Tabla 3).
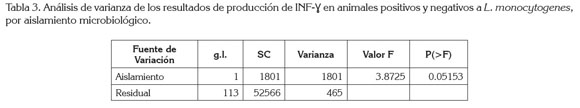
Los resultados de esta investigación demostraron, que los animales que presentaron cultivos positivos a L. monocytogenes producían cantidades bajas de IFN-γ con respecto a los animales con cultivos negativos al microorganismo; esto podría significar que aquellos animales con baja producción de IFN-γ estaban iniciando la infección y que los linfocitos T citotóxicos, T ayudadores y natural killer aun no habían producido suficiente IFN-γ para controlar la replicación y la diseminación del microorganismo, mientras que la producción alta de IFN-γ en animales negativos a cultivo son individuos cuyo sistema inmunitario logró controlar y eliminar la bacteria y, por lo tanto, en el momento de la toma de la muestra, ya no era encontrada. Estos hallazgos indican que para poder determinar si un animal es verdaderamente negativo mediante la prueba de IFN-γ, se deben tomar muestras pareadas y analizar la dinámica de producción de IFN-γ, a través del tiempo.
En conclusión, la técnica de IFN-γ para la detección de infecciones tempranas con L. monocytogenes en bovinos ofrece una importante herramienta diagnóstica, la cual, permitiría tomar decisiones profilácticas y terapéuticas en los hatos bovinos, de manera rápida y asertiva; asimismo, permitiría iniciar estudios epidemiológicos sobre la enfermedad en los hatos bovinos colombianos.
Conflictos de intereses: El manuscrito fue preparado y revisado con la participación de todos los autores, quienes declaramos que no existe ningún conflicto de intereses que ponga en riesgo la validez de los resultados presentados. Financiación: Este estudio fue financiado por la Universidad de Ciencias Aplicadas y Ambientales U.D.C.A y por la Fundación Colombiana de Estudios de Parásitos.
BIBLIOGRAFÍA
1. BAETZ, A.; WESLEY, I.; STEVENS, M. 1996. The use of listeriolysin O in an ELISA, skin test and a lymphocyte blastogenesis assay on sheep experimentally infected with Listeria monocytogenes, Listeria ivanovii or Listeria innocua. Vet. Microbiol. (Netherlands). 51:151-159.
2. BIO-X DIAGNOSTICS. 2008. Bio-X ELISA Kit for the quantitative of bovine gamma interferon (BIO K 093). (Quantitative sandwich test). Disponible desde Internet en: www.teknokroma.com (con acceso12/04/14).
3. BORUCKI, M.; PEPPIN, J. 2003. Variation in biofilm formation among strains of Listeria monocytogenes. Appl. Environ. Microbiol. (USA). 69(12):7336-7342.
4. BUBONJA, M.; WRABER, B.; BRUMINI, G.; GOBIN, I.; VELJKOVIC, D.; ABRAM, M. 2006. Systemic and local CC chemokines production in a murine model of Listeria monocytogenes infection. Mediators. Inflamm. (USA) 3:54202.
5. CASTRO, I. 2010. Asociación de ganaderos deFacatativa. Referencia Personal.
6. CHO, S. 2007. Current issues on molecular and immunological diagnosis of tuberculosis. Yonsei. Med. J. (Korea). 48(3):347-359.
7. CZUPRYNSKI, C. 2004. Listeria. In: Pathogenesis of bacterial infections in animals. C.L.; Prescott, J.F.; Songer, J.G.; Thoen, C.O. (eds.) 3a Ed. Blackwell Publishing (USA) 99p.
8. CZUPRYNSKI, C.; KATHARIOU, S.; POULSEN, K. 2010. Listeria. En: Pathogenesis of bacterial infections in animals. Gyles, C.L.; Prescott, J.F.; Songer, J.G.; Thoen, C.O. (Eds.) 4a Ed. Wiley-Blackwell (USA) 167p.
9. DHEDA, K.; POORAN, A.; PAI, M.; MILLER, R.; LESLEY, K.; BOOTH, H.; SCOTT, G.; AKBAR, A.; ZUMLA, A.; ROOK, G. 2007. Interpretation of Mycobacterium tuberculosis antigen-specific IFN release assays (T-SPOT.TB) and factors that may modulate test results. J. Infect. (USA) 55:169-173.
10. DOMÍNGUEZ, R.L.; VAZQUEZ-BOLAND, J.A.; FERNANDEZ, J.F.; ECHALECU, T.P.; GOMEZ- LUCIA, E.; RODRIGUEZ, F.E.F.; SUAREZ, F.G. 1986. Microplate technique to determine hemolytic active routine typing of Listeria strains. J. Clin. Microbiol. (USA) 24(1):99-103.
11. ELEZEBETH, S.; MALIK, S.; CHAUDHARI, S.B.; BARBUDDHE, S.B. 2007. The occurrence of Listeria species and antibodies against listeriolysin-O in naturally infected goats. Small Ruminant Res. (USA)67(2-3):173-178.
12. EPIINFO TM3.5.4. 2014. Database and Static Software; Public Health Profesional org. Center for Disease control and Prevention. (Disponible desde internet en www.cdc.gov/epiinfo/7/ (con acceso 13/10/ 2014).
13. GALLEGO, M.; TORRES, O.; SOTO, Y.; DUQUE, D.C.; BENITEZ, C. 2003. Determinación de portadores de Listeria spp. en un conglomerado lechero de la vereda Puente de Piedra del municipio de Madrid (Cundinamarca, Colombia) Rev. U.D.C.A. Act. & Div. Cient. (Colombia). 6(1):49-56.
14. GALLEGO, M.; MANRIQUE, P.; TORRES, O.; RAMÍREZ, M. 2007. Listeria monocytogenes en canales de ganado Holstein en una planta de sacrificio de la Sabana de Bogotá. Rev. U.D.C.A. Act. & Div. Cient. 8(2):95-101.
15. GASANOV, U.; HUGHES, D.; HANSBRO, P.M. 2005. Methods for the isolation and identification of Listeria spp. and Listeria monocytogenes: a review. FEMS Microbiol. Rev. (England) 29:852.875.
16. GEGINAT, G.; NICHTERLEIN, T.; KRETSCHMAR, M.; SCHENK, S.; HOF, H.; LALIC, M.; MULTHALER, M.; GOEBEL, W.; BUBERT, A. 1999. Enhancement of the Listeria monocytogenes p60 - Specific CD4 and CD8 T Cell memory by Nonpathogenic Listeria innocua. J. Inmunol. (Alemania). 162:4781-4789.
17. HANSTED, E.; SITKAUSKIENE, B.; KEVALAS, R.; TATTERSALL, A.; DAY, T. 2007. Research for practice: a new in vitro test for identification of tuberculosis infection. Medicina (Kaunas). (USA) 43 (7):519-522.
18. KAMM, C.; SKOBERNE, M.; GEGINAT, G. 2003. CD8 T cell immunome analysis of Listeria monocytogenes. FEMS Immunol. Med. Microbiol. (USA). 35(3):235-242.
19. KIM, D.; REILLY, A.; LAWRENCE, D.A. 2001. Relationships between IFNgamma, IL-6, corticosterone and Listeria monocytogenes pathogenesis inBALB/c mice. Cell. Inmunol. (USA). 207(1):13-18.
20. KISH, L. 1972. Muestreo de Encuestas. Edit. Trillas. 1°Ed. En Español. México D.F. 739p.
21. KUEHL, R.O. 2001. Diseño de Experimentos. Ed. Thomson México D.F. 666p.
22. LÓPEZ-EXPÓSITO, I.; MANSO, M.; LÓPEZ-FANDIÑO, R.; RECIO, I. 2008. Activity against Listeria monocytogenes of human milk during lactation. A preliminary study. J. Dairy Res. (England). 75:24-29.
23. LING, W.; HANHONG, D.; MIN, L. 2008. InlA and InlC2 of Listeria monocytogenes Serotype 4b Are Two Internalin proteins eliciting humoral Immune Responses Common to Listerial Infection of Various host species. Curr. Microbiol. (USA). 56:505-509.
24. LUCA, J. 2005. 3MTM PetrifilmTM Placas de Listeria. Departamento de Microbiología 3m. España.
25. MANUAL DE LA OIE. 2004. OIE., Listeria monocytogenes. Disponible desde Internet en: http://www.oie.int/esp/normes/mmanual/pdf_es/2.10.14_Listeria_monocytogenes.pdf (con acceso 04/07/2013).
26. MADARIAGA, M.; JALALI, Z.; SWINDELLS, S. 2007. Clinical utility of interferon gamma assay in the diagnosis of tuberculosis. Jabem. 20(6):540-547.
27. MOLTZ, A.; MARTIN, S. 2005 Formation of biofilms by Listeria monocytogenes under various growth conditions. J. Food. Prot. (USA). 68(1):92-97.
28. MORENO, V.F.C.; RODRÍGUEZ, M.J.; MÉNDEZ, M.M.V.M.; OSUNA, A.L.E.; VARGAS, M.R. 2007 Análisis microbiológico y su relación con la calidad higiénica y sanitaria de la leche producida en la región del Alto de Chicamocha (departamento de Boyacá). Rev. Med. Vet. Universidad de la Salle. 14:61-83.
29. NAKANE, A.; NUMATA, A.; ASANO, M.; KOHANAWA, M.; CHEN, Y.; MINAGAWA. T. 1990. Evidence that endogenous Gamma Interferon is produced early in Listeria monocytogenes infection. Infect. Immun. (Japon). 58(7):2386-2388.
30. PAILLARD, D.; DUBOIS, V. 2003. Rapid identification of Listeria species by using restriction fragment length polymorphism of PCR-amplified 23 rRNA gene fragments. Appl. Environ. Microbiol. 69(11):6386-6392.
31. PÉREZ-TRALLERO, E.; ZIGORRAGA, C.; ARTIEGA, J.; ALKORTA, M.; MARIMÓN, J.M. 2014. Two outbreaks of Listeria monocytogenes, northern Spain. Emerg. Inf. Dis. (USA) 20(12):2155-2157.
32. PINEDA, Y.; MORA, Y. 2006. Listeriosis. Revista Digital CENIAP (Colombia). No 11: mayo-agosto.
33. R CORE TEAM. 2014. R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL http://www.R-project.org/ (con acceso 08/11/2013).
34. SIERRA, S.C.; POUTOU, R.; CARRASCAL, A.K.; TORRES, K.; MERCADO, M. 2004. Validación de PCR para detección de Listeria monocytogenes en quesos frescos. Rev. U.D.C.A. Act. & Div. Cient. 7(2):53- 65.
35. SKOBERNE, M.; HOLTAPPELS, R.; HOF, H.; GEGINAT, G. 2001. Dynamic antigen presentation patterns of Listeria monocytogenes-derived CD8 T cell epitopes in vivo. J. Inmunol. 167:2209-2218
36. SKOBERNE, M.; GEGINAT, G. 2002. Efficient in vivo presentation of Listeria monocytogenes- derived CD4 and CD8 T cell epitopes in the absence of IFN-. J. Inmunol. 168: 1854-1860.
37. TIENUNGOON, S.; RATKOWSKY, D.; McMEEKIN, T.; ROSS, T. 2000. Growth limits of Listeria monocytogenes as a function of temperature, pH, NaCl and lactic acid. Appl. Environ. Microbiol.66(4):709-725.
38. TORRES, K.; SIERRA, S.; POUTOU, R.; CARRASCAL, A.; MERCADO, M. 2005 Patogenesis de la Listeteria monocytogenes. Microorganismo Zoonótico Emergente. Rev. MVZ Córdoba (Colombia).10(1):511-543.
39. VANEGAS, M.C.; MARTÍNEZ, A.J.; BOTINA, B.L. 2006. Detección por PCR de Listeria monocytogenes en productos cárnicos distribuidos en Bogotá. Rev. U.D.C.A. Act. & Div. Cient. 9 (2):149-156.
40. VERA, A.; GONZÁLEZ, G.; DOMÍNGUEZ, M.; BELLO, H. 2013. Principales factores de virulencia de Listeria monocytogenes y su regulación. Rev. Chilena Infect. 30(4):407-416.
41. VILLAMIL, D.; GALLEGO, M.M.I.; TORRES, O.A.; RAMÍREZ, M.F. 2007. Listeria monocytogenes en canales de bovinos cebú en una planta de sacrificio de la Sabana de Bogotá (Colombia). Rev. U.D.C.A Act. & Div. Cient. 10(1):93-101.
42. WILLIAMS, M.; SCHMIDT, R.; LENZ, L. 2012. Early events regulating immunity and pathogenesis during Listeria monocytogenes infection. Trends Immunol. England. 33(10):488:495.
43. ZENEWICZ, L.; SHEN, H. 2007. Innate and adaptive immune responses Listeria monocytogenes: a short overview. Microb. Infect. (France) 9:1208-1205.
Recibido: Julio 15 de 2013 Aceptado: Mayo 4 de 2015

Revista U.D.C.A Actualidad & Divulgación Científica por Universidad de Ciencias Aplicadas y Ambientales se distribuye bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.