CIENCIAS AGROPECUARIAS Y BIOLÓGICAS - Artículo Cient?fico
USO DE MICROSATÉLITES PARA LA CARACTERIZACIÓN MOLECULAR DE PALMA DE ACEITE TIPO DURA
USING MICROSATELLITES FOR MOLECULAR CHARACTERISATION OF DURA OIL PALM
Pedro Jesús Rocha Salavarrieta1; Sandra Rubiela Suárez González2; Leonardo Rey Bolívar3
1Biólogo, Ph.D., Investigador Titular, Director Laboratorio de Caracterización Molecular, Cenipalma. Dirección para correspondencia: Calle 21 No. 42C-47, Bogotá. Correo electrónico:pedro.rocha@cenipalma.org
2Química Industrial, Asistente de Investigación, Laboratorio de Caracterización Molecular, Cenipalma.
3Ingeniero Agrónomo, M.Sc., Director División de Variedades, Cenipalma.
Rev. U.D.C.A Act. & Div. Cient. 10 (2): 51-63, 2007
RESUMEN
La palma de aceite llegó a Colombia hacia la década de 1940, con materiales de siembra tipo Dura. Los cruces realizados generaron progenies que fueron establecidas en las diferentes zonas productoras del país. Sin embargo, después de varias décadas, se reconoce que la utilización de dichos genotipos podría haber sido mayor. En consecuencia, Cenipalma estableció actividades conducentes a identificar la mayoría de tales materiales Dura y, sobre lo que se encontró, realizar evaluaciones moleculares para conocer la variabilidad genética de las poblaciones y las relaciones entre los individuos, con el fin de recuperar poblaciones y emplear algunos de los individuos disponibles, como parentales en cruces controlados. Así, en el presente artículo, se reportan los resultados de la caracterización molecular, mediante la técnica de amplificación de loci microsatélites, de 80 individuos, correspondientes a 13 poblaciones de palma de aceite tipo Dura. Se concluye que, con base en su similitud genética, dichas poblaciones pueden ser clasificadas en dos grandes grupos y que es posible reconstruir en gran medida las poblaciones originales con los materiales actuales. Además, se encontró que la variabilidad genética entre las poblaciones (interpoblacional) es mayor que la encontrada dentro de cada población (intrapoblacional).
Palabras clave: Elaeis guineensis Jacq., análisis poblacional, PCR.
SUMMARY
The oil palm arrived in Colombia by the 1940?s, with the Dura type as planting material. Breeding-crosses generated progenies that were established in the different oil palm growing regions of the country. However, after several decades, it has been recognized that the use of these genotypes could have been broader. In consequence, Cenipalma established research activities to identify the maximum possible number of such Dura material and, on the resulting palms, to carry out molecular evaluations in order to determine the genetic variability of these populations and the relationship among individuals. This information will be useful in order to renew oil palm populations and to use some of these materials as parental lines in controlled crosses. Thus, in this paper, results are reported on the molecular characterization, using microsatellite-PCR-based technique, of 80 Dura-type oil palm individuals, from 13 populations. Results show that, based on their genetic similarity, these populations can be classified into two major groups and it is possible to recover, in a considerable way, original oil palm populations by using the currently identified materials. In addition, a higher interpopulation genetic variability than intrapopulation variation was found.
Key words: Elaeis guineensis Jacq., population analysis, PCR.
INTRODUCCIÓN
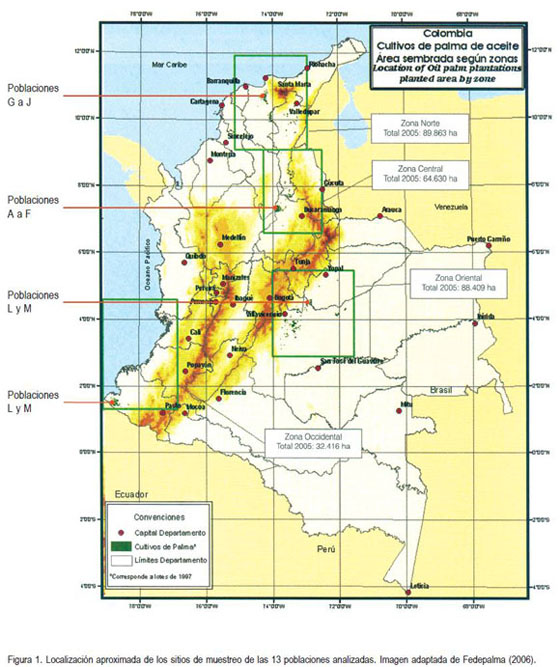
El cultivo de la palma de aceite o palma africana, Elaeis guineensis Jacq., es el que mayor cantidad de aceite produce por unidad de área cuando se compara con otras oleaginosas, tales como soya, colza, maíz y girasol (Corley & Tinker, 2003). En Colombia, desde hace ya varias décadas, la organización e institucionalidad de los cultivadores de palma aceite, representada por Fedepalma, ha hecho posible que este cultivo se convierta en una opción de desarrollo económico y social en cuatro grandes regiones, que incluyen 26 municipios de doce departamentos (Fedepalma, 2006): zona norte (Antioquia, Bolívar, Cesar, Magdalena), zona central (Cesar, Norte de Santander, Santander), zona oriental (Caquetá, Casanare, Cundinamarca y Meta) y zona occidental (Nari?o).
Cada una de las zonas palmeras colombianas posee enormes contrastes en cuanto a condiciones edafo - climáticas y de manejo agronómico, incidencia de plagas y de enfermedades y materiales genéticos, entre otras. Por esta razón, el Centro de Investigación en Palma de Aceite (Cenipalma) ha desarrollado un programa de mejoramiento genético de E. guineensis, con el fin de obtener materiales adaptados a las diversas condiciones ambientales de Colombia y con tolerancia a diferentes tipos de estrés biótico y abiótico (Rey et al. 2004).
Tres tipos de palma de aceite, Dura (D), Pisífera (P) y Ténera (T o DxP), se reconocen en estado natural con base en la presencia o ausencia de la cubierta (cuesco) de la nuez del fruto. Dicha característica está controlada por un gen mayor conocido como Sh, del inglés shell (Beirnaert & Vanderweyen, 1941; Corley & Tinker, 2003). El tipo Dura, genotipo homocigótico (Sh+/Sh+), produce un fruto con una nuez que presenta un cuesco grueso, de 2 a 8mm. El tipo Pisífera, también homocigótico (Sh-/Sh-), produce una nuez atípica que no posee cubierta o si aparece, es vestigial. El tercer material es el híbrido intraespecífico tipo Ténera, un heterocigoto (Sh+/Sh-), con cuesco de grosor intermedio, entre 0,5 y 2mm, resultado del cruce de un progenitor Dura (madre) con otro Pisífera (padre). Por presentar altos contenidos de aceite, debido a que entre el 60 y el 90% del fruto es mesocarpo (pulpa), los materiales Ténera son los más empleados comercialmente en el ámbito mundial (Corley & Tinker, 2003).
La disminución del grosor de cuesco ha sido uno de los objetivos de los programas de mejoramiento de diversas instituciones (Corley & Tinker, 2003; Rocha, 2003). Así, esta característica ha sido mapeada mediante el uso de marcadores moleculares tipo RAPD (Random Amplified Polymorphic DNA, Moretzshon et al. 2000), AFLP (Amplified Fragment Length Polymorphism, Billotte et al. 2001a) y microsatélites (Billotte et al. 2005).
El ingreso de la palma de aceite a Colombia, se inició en 1933 con material proveniente del África. Hacia 1949, se introdujeron materiales DelixDura provenientes del Asia (Bastidas et al. 2003). Uno de los aspectos más interesantes de los materiales sembrados inicialmente en Colombia y en general en el mundo, es que todos se relacionan en algún grado con cuatro palmas tipo Dura, localizadas en el jardín botánico de Bogor (Corley & Tinker, 2003). Por tal razón, es acertado considerar que la variabilidad genética que ha sido empleada en los cultivos de palma de aceite del mundo es baja. No obstante, se ha observado que en Colombia aún existen materiales tipo Dura de los que originalmente fueron introducidos al país y que se han adaptado de manera excepcional a diversas condiciones. Por esta razón, el Programa de Variedades de Cenipalma ha desarrollado una línea de investigación que permita primero conocer rápidamente la variabilidad genética de tales palmas, segundo hacer seguimiento a las características de productividad y tolerancia a plagas y enfermedades y tercero seleccionar, de manera eficiente, posibles parentales élite, con el fin de incorporarlos al programa de mejoramiento de palma de aceite. Para realizar estas actividades, el Programa ha implementado la utilización de marcadores moleculares (Rocha, 2007; 2004; 2003), en particular la detección de polimorfismos, mediante la técnica de amplificación de alelos microsatélites (Rocha et al. 2005; Montoya et al. 2005). Así, en el presente artículo, se presentan los resultados de la caracterización molecular, usando la técnica de microsatélites, de algunos de los genotipos Dura, localizados en plantaciones comerciales de las cuatro regiones palmeras colombianas, con el fin de determinar sus relaciones genéticas. Con esta información, se trata de estimar la variabilidad genética de palmas tipo Dura existentes en Colombia y, con base en las progenies comerciales actuales, a futuro, recuperar algunos de los progenitores femeninos existentes hace más de 40 a?os en Colombia.
MATERIALES Y MÉTODOS
Se muestrearon 80 palmas que representaban 13 poblaciones (figura 1). Aquí, el término ?población? hace referencia a grupos de palmas que se encontraban en distintas plantaciones y no a poblaciones naturales. Los individuos analizados corresponden a una muestra de una población de 781 individuos previamente seleccionados por caracterización morfoagronómica y cuyo índice de selección fue de 1,1 equivalente a unas 67.000 palmas iniciales.

Se recolectaron dos folíolos de hojas maduras de palmas adultas, mayores de 40 a?os de edad, los cuales se empacaron en bolsas de papel, fueron mantenidos a temperatura ambiente y transportados, vía terrestre, hasta el Laboratorio de Caracterización Molecular (LCM, Cenipalma, Bogotá). Una vez en el laboratorio, los folíolos fueron mantenidos a ?70°C hasta su uso.
Extracción del ADN y amplificación de microsatélites: El ADN fue extraído, según el protocolo reportado por Arias & Rocha (2004). Alícuotas de 50ng de ADN fueron empleadas como molde en una reacción de PCR de 25mL, que contenía solución tampón de PCR 1X (Tris-HCl 20mM, pH 8,4 y KCl 50mM); MgCl21,5mM; dNTP 200mM; 1 U de Taq DNA polimerasa (Invitrogen Life Technologies) y 0,2mM de cada cebador. En total, 80 muestras de ADN sirvieron como molde para la amplificación de los alelos presentes en 20 loci (según Billotte et al. 2001b). Las condiciones de amplificación incluyeron un minuto a 95°C, seguido por 35 ciclos de amplificación, cada uno de 94°C durante 30 segundos; 52°C durante 1 minuto y 72°C durante dos minutos. Un paso de extensión final durante ocho minutos a 72°C.
Separación y visualización: Los productos amplificados fueron separados mediante electroforesis en gel de poliacrilamida 6%, desnaturalizante (urea 5M), a 50°C, durante una hora y 30 minutos y 100 watios constantes de potencia (fuente de poder Power Pac 3000, BioRad). Los tama?os moleculares de las bandas, se determinaron por comparación con marcadores de peso molecular de ADN de 25pb y de 10pb (Cat. 10597-011 y 10821-015, Invitrogen). Los geles fueron te?idos con nitrato de plata, según la metodología descrita por Meléndez (2006) y Suárez (2006).
Análisis de bandas: Las bandas reveladas como resultado del proceso de tinción fueron registradas visualmente con ayuda de un transiluminador de luz blanca. Para cada locus, se determinó el número de alelos amplificados, utilizando, como referencia, patrones de amplificación reportados (Billotte et al. 2001b; Montoya et al. 2005; Suárez, 2006). Las bandas, se numeraron en orden descendente, tomando como referencia su respectivo peso molecular, y se codificaron en una matriz binaria donde ?1? indicó presencia y ?0? ausencia. Adicionalmente, se generó una matriz que tomó en consideración el peso de cada uno de los alelos registrados y se codificaron (con letras mayúsculas) los alelos presentes en cada locus. Esta matriz, se utilizó con el programa Popgene version 1.32 (Yeh & Boyle, 1997). Con éste, se obtuvieron las frecuencias alélicas para cada locus de cada población. A partir de éstas fue posible evaluar la variabilidad genética dentro de las poblaciones y la estructura genética (distribución de la variación dentro y entre poblaciones).
La variación genética dentro de las poblaciones, se estimó con los parámetros: porcentaje de loci polimórficos (P, un locus se consideró polimórfico cuando la frecuencia de su alelo más común no excedía 0,95); número medio de alelos por locus (A), número de alelos observados (Ao), número efectivo de alelos por locus (Ae), (Kimura & Crow, 1964), heterocigosidades (o diversidad genética) observada (Ho) y esperada (He) y Heterocigosidad promedio esperada (H). Para determinar la estructura poblacional, se calcularon los estadísticos F, según Wright (1951). Adicionalmente, para complementar los análisis, se estimaron las relaciones genéticas entre las poblaciones, mediante métodos de taxonomía numérica (análisis de similitud y análisis de clasificación aglomerativa con el programa NTSys versión 2.11L, Rohlf, 2000). Se estimaron las diferencias entre poblaciones, en términos de su distancia genética, mediante el empleo del índice de Nei (1972). La matriz de similitud para todas las poblaciones permitió agruparlas y construir dendrogramas empleando los métodos UPGMA (Unweighted Pair-Group Method Arithmetic Average, Sneath & Sokal, 1973) y el de vecinos cercanos (Saitou & Nei, 1987). La matriz de similaridad fue transformada a un producto en forma escalar con la función Dcenter (double center) de NTSys y, en consecuencia, sus valores y vectores Eigen pudieron ser calculados (Gower, 1966). El ordenamiento de los grupos en gráficas bidimensionales, se realizó con la función Eigen de NTSys. De igual manera, se estimaron las relaciones genéticas entre los individuos de las poblaciones, mediante los métodos de taxonomía numérica.
RESULTADOS Y DISCUSIÓN
Análisis poblacional
En el presente trabajo, se evaluaron 80 materiales de E. guineensis tipo Dura con 20 combinaciones de cebadores específicos para amplificar 20 loci microsatélites. Es de mencionar que, para conocimiento de los autores, esta es la primera vez que se reporta la caracterización molecular de materiales Dura de poblaciones presentes en Colombia.
El locus mEgCIR0134, se comportó como un doble locus, en consecuencia, se consideró como un locus adicional, obteniendo así amplificación para 21 loci. En total, se amplificaron 40 alelos (tabla 1), con tama?os entre 85 y 345 pares de bases (pb).
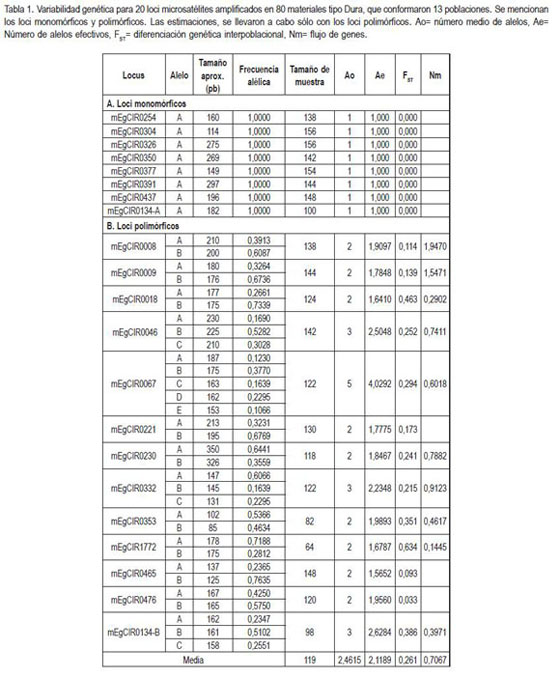
Un locus, se considera polimórfico cuando presenta más de un alelo. Para los experimentos reportados aquí, de los 21 loci amplificados, 13 (61,90%) fueron polimórficos. Un loci presentó cinco alelos, tres presentaron tres y nueve presentaron dos alelos. Para los ocho loci restantes fue posible amplificar sólo un alelo por locus. Dentro de estos, los loci mEgCIR0304 y mEgCIR0326 estuvieron presentes en el 97,5% de los individuos. Debido a la naturaleza de este estudio, relacionada con encontrar diferencias entre los materiales Dura, los ocho loci con sólo un alelo, se consideraron monomórficos y fueron eliminados para el análisis de la variabilidad genética dentro de las poblaciones (tabla 1). Es de anotar que uno de los miembros del locus doble fue monomórfico (mEgCIR0134A), mientras que el otro (mEgCIR0134B) presentó tres alelos. En total, se estudiaron las frecuencias alélicas de 32 alelos de 13 loci para las 13 poblaciones.
La media de alelos por locus polimórfico fue de 2,4615 (tabla 1), con rango de dos a cinco alelos. Estos valores son menores que los reportados por el LCM, en análisis de poblaciones E. guineensis (Montoya et al. 2005; Meléndez, 2006).
La heterocigocidad media observada para los 13 loci polimórficos fue de 0,5532; el valor más alto (0,8571) fue para el locus mEgCIR0134-B y el más bajo (0,1774) para el locus mEgCIR0018 (tabla 1).
El número medio de individuos por población fue de 6,15 (tabla 1); no obstante, las poblaciones B y H presentaron sólo tres individuos cada una, mientras que la población J incluyó a 12 individuos. Ciertamente, los tama?os de población empleados fueron peque?os y en consecuencia los resultados de los cálculos, elaborados en términos de probabilidades, pueden estar sesgados por el azar. Sin embargo, debido a la imposibilidad de contar con más individuos, pues la mayoría de las palmas de este tipo han sido erradicadas y conociendo que las palmas empleadas en este estudio pertenecían a las poblaciones iniciales o tenían algún grado de parentesco con los materiales erradicados, la información de variabilidad genética generada y las posibles relaciones entre palmas reportadas aquí, se constituyen en una herramienta de utilidad para el objetivo de recomponer poblaciones Dura colombianas.
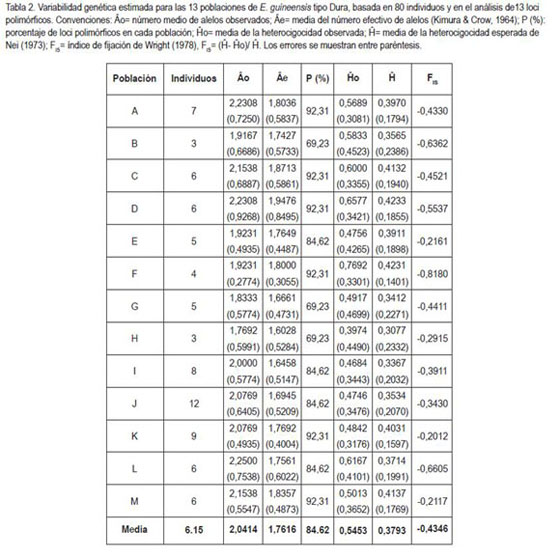
El número medio de alelos diferentes observados en cada población estuvo entre 1,7692 (población H) y 2,2500 (población L). El número medio de alelos efectivos fue de 1,7616, con rango entre 1,6028 (población H) y 1,9476 (población D). Dentro de las 13 poblaciones evaluadas, el polimorfismo medio de los loci fue de 84,62% (tabla 2).
La diversidad genética o heterocigocidad media observada (Ho) fue en promedio de 0,5453 (tabla 2), con rango entre 0,3974 (población H) y 0,7692 (población F). Estos valores son realmente altos e indican, por ejemplo, que para la población F, el 76,92% de las plantas individuales analizadas fueron heterocigotas para cualquiera de los loci analizados. Por su parte, la heterocigocidad media esperada (H) según Nei (1973) fue de 0,3793, con el valor mínimo (0,3077) para la población H y el máximo (0,4233), para la población D. Este valor es menor que el reportado por Billotte et al. (2001b) para E. guineensis (He=0,68) y, de igual manera, es menor que los valores encontrados, por el LCM, para poblaciones provenientes de la República de Angola (H= 0,515, Rocha et al. 2007).
Los valores de índice de fijación de Wright (1951) de las poblaciones analizadas, se presentaron en un rango entre ?0,2012 (población K) y ?0,8180 (población F), media de ?0,4346 (tabla 2). Los valores negativos obtenidos para todas las poblaciones (tabla 2) muestran una fuerte tendencia hacia la heterocigocidad (FIS tiene rango que va entre ?1 que indica exceso de heterocigotos hasta +1 que refleja exceso de homocigotos). Por su parte, el valor medio de la diferenciación genética interpoblacional (FST) fue de 0,261 (tabla 2). Según los rangos de interpretación planteados por Wright (1978), este valor está entre 0,25 y 1,00 e indicaría una diferenciación genética muy grande o dicho de otra manera, muestra que el 26% de la variación total, se justifica por la diferencia entre poblaciones. Sin embargo, este valor debe ser tratado con cautela, porque el rango en que se basa dicho promedio incluye valores que pasan por poca, moderada, gran y muy grande diferenciación genética (tabla 1). De todas maneras, la información obtenida permite hacer varias consideraciones. La primera, que el cruce inicial de las palmas tipo Dura (hace ya varias décadas) fue controlado y exitoso en varios casos, llevando a una diferenciación genética muy significativa entre las poblaciones del mismo tipo de material debida a una heterocigocidad alta (Ho=0.5453, tabla 2). La segunda, cuestiona la influencia del número peque?o de individuos por población en el valor real para FST. Se considera una ?población grande?, aquella en la que el número de individuos adultos es del orden de cientos más que de decenas (Falconer, 1981). La tercera consideración hace referencia a la existencia de diversos factores ambientales que pueden estar afectando el comportamiento de los genotipos, lo cual refleja ganancia genética por adaptación, lo cual es de utilidad para un programa de mejoramiento (Rey et al. 2004).
El valor medio de flujo de genes (Nm) fue de 0,7067 (tabla 1); sin embargo, el rango incluye valores que van desde 0,1445 hasta 7,4286, lo cual no permite hacer inferencias sobre tasas de evolución o fijación de alelos en cada población.
Relaciones entre poblaciones
Las relaciones entre poblaciones, se ajustan a lo esperado con base en la información histórica y de campo (figura 2). Al emplear herramientas de análisis filogenético (agrupamientos de vecinos cercanos, Saitou & Nei, 1987) se aprecian grupos de poblaciones (figura 2, los cuales se mantiene independientemente del análisis de agrupamiento u ordenación a que se sometan los datos (figura 2A, B y C). Al analizar el grupo conformado por las poblaciones G, H, I y J (cuyos valores de diversidad genética son muy similares, tabla 2) es evidente que se relacionan fuertemente entre sí. Ninguna de las poblaciones explica la variabilidad completa del grupo; pero, es claro que la recombinación que precedió a estos materiales cobijó la totalidad del grupo y, en consecuencia, el grupo se acerca a lo que debiera representar una réplica de la población inicial.
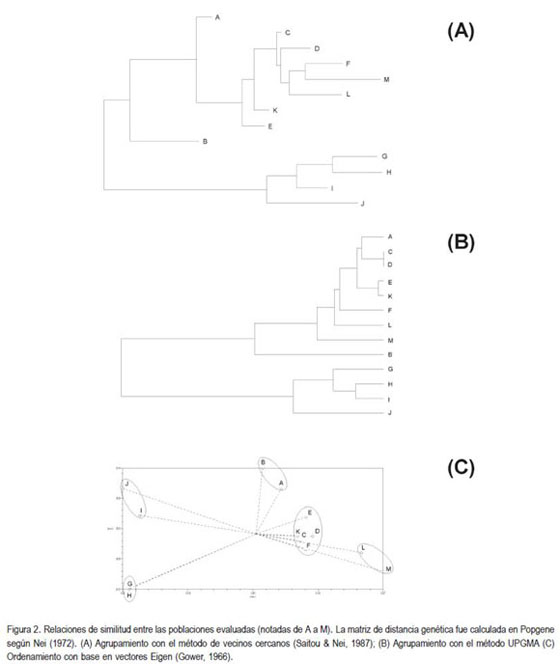
La población B está ubicada en la base de la formación de agrupamientos (figura 2A), posiblemente ésta mayor diferencia genética se relacione con progenitores que no existen en la actualidad o que no fueron muestreados. De todas maneras, los tres individuos que conforman esta población, se constituyen en entidades que pueden ayudar a restaurar poblaciones iniciales que no fueron muestreadas.
El análisis vectorial (figura 2C) muestra que las poblaciones G y H están altamente relacionadas y éstas a su vez con las poblaciones I y J. Por otra parte, las poblaciones C, D, F y K están muy relacionadas entre sí. Las poblaciones L y M y A y B se agrupan aparte (figura 2C). De nuevo, cabe anotar que estas ?poblaciones? no son poblaciones naturales, sino el resultado de una distribución aleatoria de semilla, lo cual se ve reflejado en los análisis de relaciones entre individuos.
Análisis entre individuos
Con el fin de establecer las relaciones existentes entre los individuos de las diferentes poblaciones analizadas, se llevaron a cabo estimaciones de similitud, de agrupamiento y de ordenamiento (figura 3). Se escogió el índice de Nei (1972) debido a la condición diploide de la palma y a la característica codominante de los microsatélites.
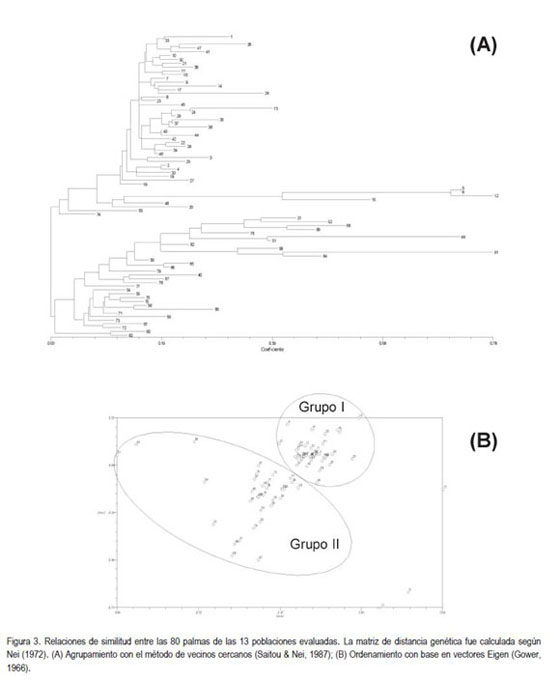
Al analizar el agrupamiento basado en el método de vecinos cercanos, se aprecia la formación de dos grandes grupos (figura 3A), los cuales están presentes también en el ordenamiento con base en vectores Eigen (figura 3B). Uno de los grandes grupos (Grupo I) está conformado por 38 de los 43 individuos de las poblaciones A, B, C, D, E, F, L y M. Tres de los cuatro individuos restantes (identificados como 6, 12 y 15) salen del grupo, debido a su alta diferenciación, la cual hace que, aún compartiendo información, probablemente derivada de parentales comunes, sus diferencias genéticas sean considerablemente altas. Los individuos 31 y 40 salen del Grupo I, debido a su similitud con individuos del Grupo II. El Grupo II está conformado por 27 de los 28 individuos de las poblaciones G, H, I y J (acorde con lo reportado en figura 2C). Debido a su similaridad con el Grupo I, el individuo restante (53) sale del Grupo II. Así, el comportamiento de las poblaciones (figura 2C) y de los individuos (figura 3) es bastante similar. Sin embargo, la única población que aparentemente no se ajusta a las distribuciones en grupos es la población K. Esta población (de nueve individuos), se agrupa en el análisis por poblaciones con las poblaciones C, D, E y F (figura 2 A y C). Sin embargo, el análisis por individuos (figura 3B) muestra que dicha ?población? tiene individuos que se distribuyen entre los dos grupos encontrados, seis individuos (44 a 49) en el Grupo I y sólo tres individuos (50 a 52) en el Grupo II. Evidentemente, la contribución del ordenamiento se ve muy afectada por el tama?o de la población.
CONCLUSIONES
El éxito de un programa de fitomejoramiento está en la disponibilidad de material parental y de sistemas de selección eficientes. Con el objeto de determinar la variabilidad genética de los materiales tipo Dura y generar información de utilidad para una posible recomposición de poblaciones, se obtuvo información de 13 loci microsatélites (de 20 evaluados), los cuales fueron bastante eficientes e informativos (tabla 1, sección B).
Se mostró una diferenciación genética alta. En particular, se determinó que la variabilidad entre las poblaciones de materiales tipo Dura analizadas es mayor que la variabilidad genética hallada dentro de cada una de las poblaciones. Además, los análisis de relaciones genéticas entre individuos de las distintas poblaciones agruparon a la mayoría de individuos y de poblaciones en dos grupos. Se identificaron aquellos individuos (6, 12 y 15) que salen de los agrupamientos y otros que por su similitud podrían ser seleccionados como representantes de cada población o grupo.
Así, la información obtenida en el presente estudio es importante para el programa de fitomejoramiento de Cenipalma, debido a que se ha ampliado el conocimiento genético de materiales que, aparte de tener características morfoagronómicas de interés (tolerancia a estrés biótico y abiótico) y de haber ganado adaptación a distintas condiciones medioambientales, fueron utilizados exitosamente en el pasado, pero no se conservaron para los futuros programas de fitomejoramiento.
Por su variabilidad genética, características morfoagronómicas y de adaptación, los materiales evaluados se constituyen en un elemento importante para el rescate y la generación de combinaciones deseables de genes que serán de utilidad para el desarrollo de la palmicultura colombiana.
AGRADECIMIENTOS
Los autores agradecen al ingeniero Iván Ayala (Cenipalma) por el suministro de material biológico, al Comité Editorial de Cenipalma, por la crítica de este manuscrito y a los árbitros externos por sus valiosos comentarios. Esta investigación fue patrocinada por el Fondo de Fomento Palmero (FFP), administrado por Fedepalma.
BIBLIOGRAFÍA
1. ARIAS, D.; ROCHA, P. 2004. Análisis de diversidad genética en materiales tolerantes y susceptibles a la pudrición de cogollo en palma de aceite mediante marcadores moleculares. Palmas (Colombia). 25(3):11-27.
2. BASTIDAS, S.; PE?A, E.; REYES, R. 2003. Genealogía del germoplasma de palma de aceite (Elaeis guineensis Jacq.) del proyecto de mejoramiento genético de Corpoica. Palmas. 24(1):21-29.
3. BEIRNAERT, A.; VANDERWEYEN, R. 1941. Contribution a l?étude g?nétique et biométrique des variétés d? Elaeis guineensis Jacq. Publ. Inst. Natl. Etude Agron. Congo Belge, Ser. Sci. (Belgium). 27:1-101.
4. BILLOTTE, N.; FRANCES, L; AMBLARD, P.; DURAND-GASSELIN, T. RUSTERUCCI, A.M.; NOYER, J.L.; COURTOIS, B. 2001a. Search for AFLP and microsatellite molecular markers of the SH gene in oil palm (Elaeis guineensis Jacq.) by bulk segregant analysis (BSA) and by genetic mapping. Proceedings of 2001 PIPOC International Palm Oil Congress ? Cutting-edge technologies for sustained competitiveness (Agriculture). Malaysia. p.442-445.
5. BILLOTTE, N.; RUSTERUCCI, A.M.; BARCELOS, E.; NOYER, J.L.; AMBLARD, P.; BAURENS, F.C. 2001b. Development, characterization, and across-taxa utility of oil palm (Elaeis guineensis Jacq.) microsatellite markers. Genome (Canada). 44(3):413-425.
6. BILLOTTE, N; MARSEILLAC, N; RISTERUCCI, A.M; ADON; B; BROTTIER, P.; BAURENS, F.C.; SINGH, R.; HERRÁN, A.; ASMADY, H.; BILLOT, C.; AMBLARD, P.; DURAN-GASSELIN, T.; COURTOIS, B.; ASMONO, D.; CHEAH, S.C.; ROHDE, W.; RITTER, E.; CHARRIER, A. 2005. Microsatellite-based high density linkage map in oil palm (Elaeis guineensis Jacq.). Theoret. Appl. Genet. (Germany). 110:754-765.
7. CORLEY, R.H.V.; TINKER, P.B. 2003. The oil palm. 4th ed. Blackwell Publ. Great Britain. 562p.
8. FALCONER, D.S. 1981. Introduction to quantitative genetics. 2nd ed. Longman. New York. 340p.
9. FEDEPALMA. 2006. Statistical yearbook 2006. The oil palm agroindustry in Colombia and the World 2001-2005. Fedepalma, Bogotá, 122p.
10. GOWER, J.C. 1966. Some distance properties of latent root and vector methods used in multivariate analysis. Biometrika (United Kingdom). 53:325-338.
11. KIMURA, M.; CROW, J.F. 1964. The number of alleles that can be maintained in a finite population. Genetics (U.S.A.). 49:725-738.
12. MELÉNDEZ, E. 2006. Caracterización molecular de palma de aceite, Elaeis guineensis Jacq., procedente de Angola mediante microsatélites en Cenipalma. Trabajo de Grado. Universidad Francisco de Paula Santander, Cúcuta. 93p.
13. MONTOYA, C.; ARIAS, D.; REY, L.; ROCHA, P.J. 2005. Diversidad genética de materiales Elaeis guineensis Jacq. procedentes de Angola. Fitotecnia Colombiana. 5(2):1-10.
14. MORETZSOHN, M.C.; NUNES, C.D.M.; FERREIRA, M.F.; GRATTAPAGLIA D. 2000. RAPD linkage mapping of the shell thickness locus in oil palm (Elaeis guineensis Jacq.). Theoret. Appl. Genet. 100:63-70.
15. NEI, M. 1972. Genetic distance between populations. Am. Nat. 106:283-292.
16. NEI, M. 1973. Analysis of gene diversity in subdivided populations. Proc. Nat. Acad. Scienc. 70:3321-3323.
17. REY L.; GÓMEZ P.L.; AYALA I.; DELGADO W.; ROCHA, P. 2004. Colecciones genéticas de palma de aceite Elaeis guineensis (Jacq.) y Elaeis oleifera (H.B.K) de Cenipalma: características de importancia para el sector palmicultor. Palmas. 2(No. Especial):39-48.
18. ROCHA, P.J. 2003. Marcadores moleculares, una herramienta útil para la selección de palma de aceite. Palmas. 24(2):11-25.
19. ROCHA, P.J. 2004. Conceptos básicos en biotecnología de la palma de aceite. Palmas. 25(No. Especial. Tomo 1.):1-17.
20. ROCHA, P.J. 2007. La biotecnología como una herramienta para agilizar la selección de nuevos materiales en palma de aceite. Palmas.28(No. Especial. Tomo 1): 144-148.
21. ROCHA, P.J.; MELÉNDEZ, E.; REY, L. 2007. Ampliación del análisis de diversidad genética de palma de aceite proveniente de Angola. Respuestas (Colombia). 12(1):20-28.
22. ROCHA, P.J.; ROJAS, Y.; REY, L. 2005. Caracterización molecular preliminar del banco de germoplasma de Elaeis oleifera [H.B.K.] Cortés, mediante microsatélites. Ceniavances (Colombia). No. 130:1-4.
23. ROHLF, F.J. 2000. NTSYSpc: Numerical taxonomy and multivariate system. Version 2.1. Exeter Publ. Ltd. Setauket, N.Y. Programa informático.
24. SAITOU, N.; NEI, M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. and Evol. (United Kingdom). 4:406-425.
25. SNEATH, P.H.A.; SOKAL, R.R. 1973. Numerical taxonomy. Freeman, San Francisco, CA. 573p.
26. SUÁREZ, S.R. 2006. Estudio preliminar de la caracterización molecular por microsatélites de progenitores tipo Dura de palma de aceite (Elaeis guineensis Jacq.). Trabajo de Grado. Facultad de Química Industrial. Universidad de Ciencias Aplicadas y Ambientales (U.D.C.A) y Corporación Tecnológica de Bogotá (C.T.B.). Bogotá. 90p.
27. WRIGHT, S. 1978. Evolution and the genetics of populations. Vol. 4. Variability within and among natural populations. University of Chicago Press, Chicago, IL. 581p.
28. WRIGHT, S. 1951. The genetical structure of populations. Ann. Eugenics (United Kingdom). 15:323-354.
29. YEH, F.C.; BOYLE, T. 1997. Population genetic analysis of co-dominant and dominant markers and quantitative traits. Belgian J. Bot. (Belgium) 129:157.
Recibido: Marzo 15 de 2007; Aceptado: Agosto 6 de 2007

Revista U.D.C.A Actualidad & Divulgación Científica por Universidad de Ciencias Aplicadas y Ambientales se distribuye bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.