CIENCIAS AGROPECUARIAS Y BIOLÓGICAS - Artículo Científico
AISLAMIENTO E IDENTIFICACIÓN DE Lactobacillus CONTAMINANTES EN UNA PLANTA COLOMBIANA DE FERMENTACIÓN ALCOHÓLICA
ISOLATION AND IDENTIFICATION OF CONTAMINANT Lactobacillus IN A COLOMBIAN ALCOHOL FERMENTATION PLANT
Diana Patricia Sossa Urrego1, Lina María González2, María Consuelo Vanegas3
1 Microbióloga Industrial, Laboratorio Ecología Microbiana y Alimentos LEMA. Facultad de Ciencias. Departamento Ciencias Biológicas. Universidad de los Andes, Bogotá D.C., Colombia Correo electrónico:dp.sossa57@uniandes.edu.co
2Microbióloga, Laboratorio Ecología Microbiana y Alimentos LEMA, Facultad de Ciencias. Departamento Ciencias Biológicas. Universidad de los Andes, Universidad de los Andes, Bogotá D.C., Colombia. Correo electrónico:magogii@gmail.com
3 Microbióloga. MSc. Ciencias Biológicas. Directora Laboratorio Ecología Microbiana y Alimentos LEMA, Facultad de Ciencias. Departamento Ciencias Biológicas. Universidad de los Andes, Universidad de los Andes, Bogotá D.C., Colombia, Cra. 1a No 18a-70 Edificio J209. Correo electrónico:mvanegas@uniandes.edu.coAutor de correspondencia.
Rev. U.D.C.A Act. & Div. Cient. 12 (2): 163-172, 2009
RESUMEN
La contaminación bacteriana es un problema continuo en procesos fermentativos, particularmente, en fermentaciones para producción de etanol, donde se utiliza melaza de caña o maíz como materia prima; estos contaminantes son bacterias ácido lácticas, predominando el género Lactobacillus, principalmente. Estas bacterias crean un descenso constante del carbono disponible en la materia prima para la conversión a etanol. Compiten por factores de crecimiento necesarios para que la levadura realice su proceso fermentativo; así mismo pueden producir ácido láctico, el cual, inhibe el crecimiento de la levadura, generando una pérdida de dos moléculas de etanol por cada molécula de ácido láctico producido, disminuyendo la productividad y ocasionando pérdidas económicas. En este estudio, se colectaron muestras en una destilería colombiana de materia prima, levadura utilizada para el inóculo y cinco tanques de fermentación a diferentes tiempos, donde las bacterias ácido lácticas contaminantes fueron cuantificadas, aisladas y caracterizadas polifásicamente, utilizando métodos fenotípicos y genotípicos. Se emplearon pruebas bioquímicas de microbiología clásica, como método fenotípico y la técnica de PCR (Reacción en Cadena de la Polimerasa), como método genotípico. Al cuantificarlas, se evaluó su comportamiento y su impacto en la eficiencia del proceso. Se observó recuentos altos de bacterias ácido lácticas en los tanques de fermentación; también se hallaron estas bacterias en la materia prima y levadura, lo que sugiere que el proceso tiene varias fuentes de contaminación. Al realizar la identificación polifásica, el género de bacterias más representativas fue Lactobacillus.
Palabras clave:Producción de etanol, contaminación bacteriana, bacterias ácido lácticas, Lactobacillus.
SUMMARY
Bacterial contamination is a constant problem in fermentation processes, especially in the production of ethanol where sugar cane or corn molasses is used as raw material. The main contaminants are lactic acid bacteria, predominating, the genus Lactobacillus. These bacteria cause a decrease in the carbon source for the ethanol production. They compete for growth factors, needed by the yeast cells to generate the fermentation process. They also produce lactic acid, an inhibitor of yeast growth, which causes the loss of two molecules of ethanol per molecule of lactic acid produced. This causes a decrease in productivity, thus originating economic losses. In this study samples were collected from a Colombian distillery, from raw material, yeast used as inoculum and from five fermentation tanks at different moments. The lactic bacteria contaminants were quantified, isolated and characterized using phenotypic and genotypic methods. Biochemical tests of classical microbiology were used as the phenotypic method and PCR (Polimerase Chain Reaction) for the genotypic method. When quantifying the bacteria, their behavior and impact in efficiency was evaluated. High counts of lactic acid bacteria were observed in fermentation tanks. These bacteria were also found in the raw matter and in the yeast, suggesting the process has several sources of contamination. When performing the polyphasic characterization, the main contaminant was Lactobacillus.
Key words:Ethanol production, microbial contamination, lactic acid bacteria, Lactobacillus.
INTRODUCCIÓN
Debido a la creciente demanda de etanol en el país y al aumento del número de destilerías para la producción de etanol en el contexto de la ley 693 de 2001, el estudio de las condiciones de producción de etanol y la optimización de proceso ha cobrado gran importancia. Dicha ley decreta que la gasolina utilizada en los centros urbanos de más de 500.000 habitantes tendrá que contener componentes oxigenados, tales como alcoholes carburantes, para generar una reducción de emisiones de CO entre 22-50%, mejorando la calidad del aire y contribuyendo a la reducción del calentamiento global, cumpliendo así con el mandato del Protocolo de Kyoto (Arias et al. 2006).
En pro de obtener mejores rendimientos en el proceso de destilación, las plantas productoras de etanol buscan obtener máxima productividad, mediante estrategias que garanticen la disminución del costo y el aumento en la eficiencia del proceso.
Se ha encontrado que las Bacterias Ácido Lácticas (BAL) son el contaminante primario de las destilerías, que constituyen un grupo de bacterias Gram positivas, no esporuladas, con morfología de cocos y de bacilos, que producen ácido láctico, como producto mayor de fermentación de carbohidratos (Lars, 2004), lo cual, genera pédidas de etanol, ya que por cada gramo de ácido láctico, se pierden 0,51g de etanol (Glazer & Nikaido, 1998). En diferentes destilerías se ha identificado que las BAL constituyen la mayoría de las bacterias contaminantes, donde Lactobacillus sp. es la especie más abundante, en un 44-60% del total de aislamiento; otras especies en baja concentración son Lactobacillus delbrueckii subsp. delbruecki, L. acidophilus, L. crispatu, Lactococcus, Leuconostoc, Pediococcus y Weisella (Skinner & Leathers, 2004).
Estas BAL reducen la eficiencia del proceso, porque tienen necesidades nutricionales parecidas a las de la levadura, produciendo una competencia por el carbono disponible (Bayrock & Ingledew, 2004) y factores de crecimiento (Narendranath et al. 1997), generando la floculación de la levadura y disminuyendo el consumo de carbohidratos, reduciendo así el etanol de 2-7% v/v (Narendranath et al. 1997) y ocasionando pérdidas, incluso, del 1% de etanol, equivalente a una perder más de 300.000 dólares en una planta que produce 100.000.000 litros de etanol por año (Hynes et al. 1997).
Los métodos utilizados en la industria para controlar esta contaminación incluyen procesos de limpieza y de desinfección (Narendranath et al. 1997), pero en contaminaciones crónicas, se puede requerir paradas de proceso, generando pérdidas económicas. Otro método de control es la adición de sustancias antimicrobianas que no tienen efecto sobre la levadura, como la penicilina G y la virginiamicina, las cuales, se venden comercialmente y en muchas destilerías utilizan estos antibióticos, como profilaxis, por su eficiente control (Hynes et al. 1997).
En las destilerías colombianas no se han estudiado estas bacterias contaminantes, aunque su análisis facilitaría las estrategias de control en los procesos de producción en las diferentes etapas de proceso, ya que éstas pueden ser transmitidas por el sustrato empleado para la fermentación, en la levadura seca utilizada para la producción del inóculo, en los tanques, en las líneas de traslado y en las materiales que se recirculan (Hynes et al. 1997). Por lo anterior, el objetivo de este estudio fue cuantificar, aislar e identificar las BAL contaminantes del proceso de producción de etanol, generando conocimiento básico para futuras investigaciones, que tengan por objetivo proponer nuevas alternativas que ayuden a optimizar el proceso y evitar pérdidas económicas en la industria.
Basados en el estudio realizado por Skinner & Leathers (2004), quienes reportan que la mayoría de estas BAL contaminantes pertenecen al género Lactobacillus y pretendiendo evaluar si en la destilería colombiana se mantenía esta conclusión, se utilizó una metodología de identificación polifásica, para el género Lactobacillus, basados en características morfológicas, bioquímicas y complementándose con datos obtenidos de técnicas moleculares, integrando así criterios fenotípicos y genotípicos, para generar una identificación polifásica.
MATERIALES Y MÉTODOS
Cepas referencia: Se utilizó, como control positivo, Lactobacillus plantarum WS417, donada por Zentralinstitut für Ernährungs und Lebensmittelforschung (Ziel), de la Universidad München en Freising Alemania y, como control negativo Listeria monocytogenes ATCC 7644. Las cepas fueron mantenidas en congelación a -20°C, en caldo Man Rogosa and Sharpe Broth MRS (Scharlau, España), con gliceron al 10% v/v, al igual que las cepas aisladas en este estudio.
Muestreo: Las cinco muestras de materia prima (miel virgen), las dos muestras de levadura seca empleadas como inóculo de fermentación y las cinco muestras provenientes de tanques de fermentación (volumen efectivo trabajo 92.000 y 180.000 litros, fermentación por lote alimentado), tomadas a diferentes tiempos (4, 10, 19, 27 y 41 horas de trabajo), fueron obtenidas de una destilería colombiana, con capacidad de producción de 60.000 litros (Gnecco, 2006). Las muestras fueron mantenidas en refrigeración 4°C±2°C hasta el momento del análisis (Dennis & Ingledew, 2005).
Consumo de sustrato en tanques de fermentación: En los tanques de fermentación, se determinaron los °Brix, utilizando un aerómetro Brix (modelo HB-80-02, Xinfeng), con rango de medición de 0-10 y escala de 0,1°Brix, como medida de sólidos disueltos, para determinar el consumo del sustrato.
Eficiencia de fermentación: Los datos del porcentaje de eficiencia de fermentación, por cada tanque, fueron suministrados por la planta productora de etanol, calculada a partir del balance de materia generado por la base de datos de la empresa.
Recuento y aislamiento de BAL: Para la medición de la carga microbiana, se realizaron diluciones seriadas de la muestra en agua peptonada al 0,1% (Oxoid, Inglaterra), que fueron sembradas en agar MRS (Scharlau, España), suplementado con cicloheximida al 0,001% (SR0222, Inglaterra) e incubadas a 30°C ± 2°C por 72 h, sin agitación y en condiciones de aerobiosis. A partir de los recuentos, se realizaron aislamientos para obtener cultivos con un solo tipo de microorganismo (Skinner & Leathers, 2004).
Identificación por microbiología tradicional: Los cultivos obtenidos fueron clasificados utilizando criterios macroscópicos, microscópicos y bioquímicos (catalasa y oxidasa), para realizar su posterior identificación (Kandler & Weiss, 1984).
Las bacterias con morfología de bacilos Gram positivos, se cultivaron a pH 9,0, para clasificarlas entre los géneros Lactobacillus y Carnobacterium (Lars, 2004). Con el propósito de realizar la identificación del género Lactobacillus, se evaluó motilidad y fermentación de azúcares, como lactosa, fructuosa, galactosa, glucosa, maltosa, manitol, manosa, sorbitol y sacarosa marca Oxoid (Kandler & Weiss, 1984) y se determinó el perfil bioquímico, consultando el manual de Bergey (Kandler & Weiss, 1984).
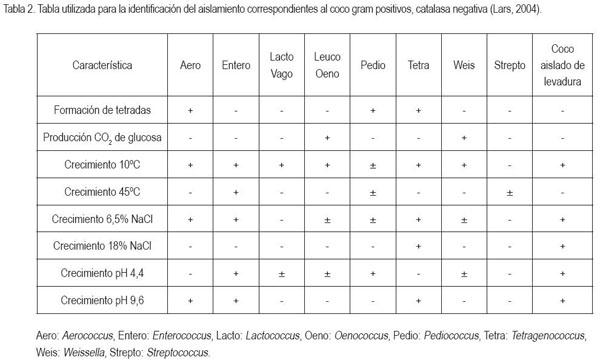
El aislamiento correspondiente a la morfología de coco Gram positivo fue clasificado entre los géneros Aerococcus, Enterococcus, Lactococcus, Leuconostoc, Pediococcus, Streptococcus y Wissella, para lo cual, se realizaron las siguientes pruebas: motilidad (Oxoid, Inglaterra), formación de tétradas, formación de CO2 a partir de glucosa, crecimiento a 10°C, crecimiento a 45°C, crecimiento a 6,5% NaCl, crecimiento a 18% NaCl, crecimiento a pH 4,4; todas las pruebas fueron realizadas en caldo MRS suplementado, dependiendo del objetivo de la prueba (Lars, 2004). La verificación, se realizo recurriendo a Gram-positive ID sistem/BD BBL CRYSTAL GP (Becton Dickinson & Company, USA).
Identificación molecular del género Lactobacillus: Para la identificación molecular de las cepas sospechosas del género Lactobacillus, se aplicó la técnica PCR, basada en el protocolo reportado por Dubernet et al. (2002), quienes recomiendan los primers LbLMA1-rev y R16-1 (Bioengland, UK); con el fin de llevar a cabo la amplificación de la Secuencia Intergénica Ribosomal (ITS) 16S/23S, se utilizó una reacción de volumen final 25µL, que contenía 1X de PCR Go-Taq green master mix (Promega Corporation, Madison, USA), 10µM de cada primer, agua libre de nucleasas y 50ng de ADN. Los productos fueron visualizados en un gel de agarosa (Merck, USA) 2%, teñido con Bromuro de Etidio; finalmente, la electroforesis, se realizó a 80V y 400mA.
Identificación polifásica del género Lactobacillus: La identificación polifásica es una estrategia confiable de identificación, como lo reporta Prakash et al. (2007), ya que permite integrar técnicas de microbiología tradicional con técnicas de biología molecular, combinando así características fenotípicas con genotípicas e integrando los resultados obtenidos des esta caracterización (Prakash et al. 2007).
RESULTADOS Y DISCUSIÓN
Recuento de BAL: En la materia prima estudiada, se obtuvo un recuento de BAL de 32x103UFC/mL, resultado coherente con la procedencia de la materia prima, que se obtiene del proceso de extracción del jugo de la caña de azúcar, la cual, contiene muchas impurezas del suelo, de donde pueden aislarse las BAL; además, a la miel no se extrajo el azúcar, lo que la hace susceptible a la contaminación, por ser un sustrato muy rico en carbono. Estos resultados demuestran que gran proporción de la materia prima empleada para el llenado de los tanques de fermentación es una fuente importante de contaminación para el proceso, como lo reporta Hynes et al. (1997), indicando que se están adicionando, constantemente, contaminantes al proceso.
La levadura manipulada en la etapa de propagación presentó un recuento de BAL de 20x101UFC/mL, generando otra fuente de contaminación. Lo anterior soporta lo enunciado por Hynes et al. (1997), quienes reportaron la presencia de BAL en la levadura usada como inóculo. El aporte microbiano por parte de la levadura al tanque de fermentación puede generar bajas eficiencias en la fermentación, de no ser controlada eficazmente.
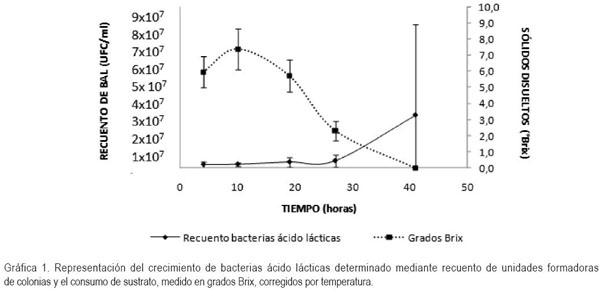
En los tanques de fermentación se obtuvieron recuentos de BAL en el orden 106UFC/mL desde el principio del proceso, como se puede observar en la gráfica 1, lo cual, puede ser el resultado de la deficiencia en las medidas de control para la contaminación en la etapa de propagación de la levadura.

A diferencia de las levaduras, las BAL no encuentran condiciones óptimas de crecimiento en altas concentraciones de azúcar (Rovira & Rovira, 1994) (Gráfica 1), donde se observa que las BAL incrementan su crecimiento después de 27 horas de trabajo, en una concentración de sólidos disueltos de 2,3°Brix, mostrando una relación inversamente proporcional entre la concentración del sustrato y el crecimiento del contaminante; lo anterior, puede ser la consecuencia del estrés osmótico causado por la alta concentración de sólidos disueltos, que al ser disminuida ofrece a las BAL condiciones excelentes de crecimiento. Este comportamiento, se relaciona con lo reportado por Rovira & Rovira (1994), quienes especifican que la materia prima diluida es más susceptible a contaminación que la materia prima pura.
Otro factor que puede aumentar el crecimiento del contaminante a las 27 horas de trabajo es la falta de dosificación de antibiótico, ya que los pulsos de antibiótico establecidos en el proceso de la destilería evaluada, solo son adicionados hasta la hora 20 (instructivo de fermentación con melaza IN-DD-15, 2006. Propiedad intelectual de la empresa productora de etanol), dejando sin protección antimicrobial la fermentación, contribuyendo al incremento de las BAL.
Los tanques de fermentación presentaron una eficiencia promedio de 80,19%, resultado que representa una pérdida 7,81% en la eficiencia del proceso de fermentación, al ser comparada con la eficiencia teórica manejada en la planta de producción (88%). Esto, se relaciona con el estudio reportado por Hynes et al. (1997), en el cual, al realizar infecciones artificiales en rangos de 105-109UFC/mL con L. plantarum, L. paracasei, L. rhamnosus y L. fermentum, se observó una reducción de etanol hasta 7,6%. También se encontró relación con lo registrado por Narendranath et al. (1997), quienes efectuaron infecciones con BAL en fermentaciones a nivel laboratorio en rangos de 106-109UFC/mL generando una reducción en el porcentaje de etanol, del 2 al 7%.
Una pérdida de eficiencia de 7,81% en un fermentador con grado alcohólico esperado de 8,0% v/v y volumen de trabajo de 92.000 litros, genera una pérdida de 554 litros de etanol al 96,3% v/v, que tiene un valor comercial promedio de $2.300 (1,18 USD), por lo tanto, las pérdidas asciendes a $1.274.200 (653,77 USD), por tanque de fermentación.
Identificación microbiología tradicional: Se aislaron 38 cepas catalasa negativa, caracterizadas microscópicamente como bacilos Gram positivos, en una proporción del 97,37% (37 cepas), siendo el porcentaje restante equivalente a cocos Gram positivos (una cepa); está última aislada de una muestra de levadura seca.
Se identificaron 20 aislamientos correspondientes al género Lactobacillus, de los cuales, cuatro fueron caracterizados hasta especie (tres cepas correspondientes a L. kandleri y una cepa a L. delbrueckii subsp. delbrueckii), logrando reconocer, en su totalidad, el 54,05% de los bacilos Gram positivos aislados (Tabla 1). El coco Gram positivo no pudo ser identificado con las pruebas fenotípicas realizadas, de acuerdo a los parámetros establecidos por Lars (2004), como se muestra en la tabla 2, por lo tanto, fue necesario emplear un método de identificación rápida, como el Gram-positive ID sistem/BD BBLCRYSTAL GP (Becton Dickinson & Company, USA), con el que se determinó la cepa como Streptococcus porcinus, con un porcentaje de identidad del 99% (software BBL Crystal MIND, Becton Dickinson & Company, USA). Este resultado no presenta concordancia con las pruebas fenotípicas utilizadas para la identificación del género (Lars, 2004) (Tabla 2), ya que las pruebas de crecimiento a 10°C, crecimiento 6,5% NaCl, crecimiento 18% NaCl, crecimiento pH 4,4 y 9,6, son negativas para el género Streptococcus, resultados no concordantes con los obtenidos por el aislamiento. Además, el S. porcinus está asociado con infecciones en cerdos, como linfadenitis cervical y con aborto, y en casos de endocarditis o microbiota de cerdos sanos (Duarte et al. 2005). El nicho de este microorganismo no está relacionado con el ambiente de una fermentación alcohólica, lo que hace poco probable el resultado evidenciado por la técnica rápida Crystal BBL. Para la identificación del coco Gram positivo es necesario realizar, por lo tanto, más pruebas bioquímicas.

La diversidad metabólica de las BAL y su habilidad para intercambiar material genético (Rixon & Warner, 2003), debido a la frecuencia de transferencia de plásmidos (Singh et al. 2008), dificulta su identificación, aplicando técnicas de microbiología tradicional, lo que implica el empleo de numerosas pruebas bioquímicas que no discriminan con exactitud entre las especies, lo que se relaciona con lo reportado por Dubernet et al. (2002) y con lo mencionado en el Manual de Bergey (Kandler & Weiss, 1984). Por lo tanto, las pruebas bioquímicas, por sí solas, no son suficientes para la diferenciación de especies.
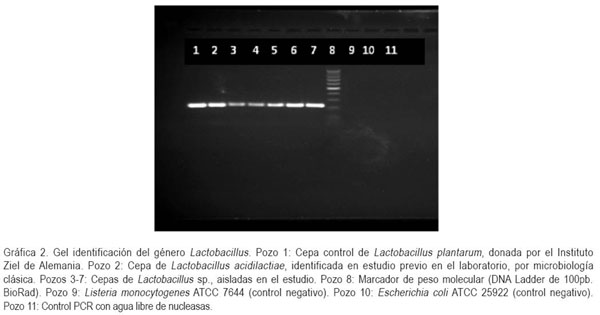
Identificación por biología molecular: Utilizando el método de biología molecular (PCR), se logró la identificación de 19 cepas, equivalentes al 50% del total de los aislamientos. Las cepas identificadas como Lactobacillus sp. presentaron un amplicon de tamaño aproximado de 250pb, igual al generado por el control positivo L. plantarum WS417 (Gráfica 2); el control negativo L. monocytogenes ATCC 7644 no presentó producto de amplificación, al igual que la cepa correspondiente a coco Gram positivo, aislada de la levadura (dato no mostrado). Estos resultados, se relacionan con el reporte de Dubernet et al. (2002), donde se determina que al utilizar en la PCR los primers LbLMA1-rev y R16-1, con el propósito de realizar la amplificación de la Secuencia Intergénica Ribosomal (ITS) 16S/23S específica para el género Lactobacillus, se obtiene una banda de 250pb, aproximadamente.
La identificación de las bacterias contaminantes del proceso de producción de etanol pertenecen al género Lactobacillus, ya sea en una proporción de 52,63% (20 cepas), del total de los aislamientos, identificadas por el método de microbiología tradicional o la proporción de 50% (19 cepas), del total de los aislamientos, identificadas por biología molecular, resultados relacionados con los reportados por Skinner & Leathers (2004), donde al analizar muestras de tanques de fermentación y líneas de traslado de una destilería, se identifican Lactobacillus sp., en una proporción 44-60%, además de un aislamiento de L. delbrueckii subsp. delbrueckii, como la encontrada en este estudio.
Identificación polifásica: Al integrar los resultados obtenidos en la caracterización fenotípica (microbiología tradicional) y genotípica (biología molecular) para realizar la identificación polifásica, se evidenció una similaridad de los resultados, en un 21,62% (ocho cepas de los aislamientos de correspondientes a bacilos Gram positivos), para los aislamientos identificados como Lactobacillus sp. (Tabla 1). Los aislamientos restantes no fueron identificados polifásicamente, ya que no se encontraron resultados similares en las pruebas fenotípicas y genotípicas; un 32,43% (doce cepas) de los aislamientos de bacilos Gram positivos fue identificado por el método fenotípico, pero no genotípico y el 29,73% (once cepas) de los aislamientos de bacilos Gram positivos, se identificó por el método genotípico y no fenotípico. Para lograr la identificación polifásica de estos aislamientos es necesario realizar más pruebas bioquímicas, como la fermentación de otros carbohidratos, hidrólisis de arginina, tolerancia a bilis, producción de polisacáridos extracelulares, tipificación serológica (Lars, 2004) o utilizar métodos rápidos, como API y otras técnicas moleculares, como ribotipificación o análisis de macrorestricción (Singh et al. 2008).
Los métodos de identificación fenotípicos y genotípicos propuestos no lograron la determinación de seis de los aislamientos, correspondientes a bacilos gram positivos, que podrían responder al género Carnobacterium (bacilos Gram positivos, catalasa negativa), pero se hace necesario, para su identificación, realizar otras pruebas bioquímicas, como formación de ácido, a partir de amigdalina, inulina, manitol, D-xilosa y una PCR específica para este género o una PCR para amplificar el 16S (Holt et al. 1994). La prueba que se tenía especificada para la clasificación de los bacilos gram positivos entre el género de Lactobacillus y Carnobacterium (crecimiento a pH 9) no fue específica, porque la cepa utilizada como control de Lactobacillus (L. plantarum) presentó crecimiento, lo cual, no debería ocurrir, según lo reportado por Lars (2004), mostrando su baja especificidad.
De los resultados anteriores, se desprende que la producción de etanol no es llevada a cabo en condiciones de un cultivo puro (Narendranath et al. 1997), lo cual, es evidenciado al cuantificar la población de BAL en las diferentes muestras analizadas de miel virgen, levadura seca y tanques de fermentación, confirmando que las BAL son contaminantes en las fermentaciones para la producción de etanol.
En los tanques de fermentación evaluados, se halló alta concentración de BAL, lo que puede ser un factor que explique la pérdida de la eficiencia de fermentación. De las BAL aisladas, se identificaron en mayor proporción bacterias del género Lactobacillus sp., en muestras de materia prima, levadura y tanques de fermentación, lo cual, demuestra que la miel virgen y la levadura son fuentes de contaminación que alimentan los tanques de fermentación. Finalmente, estos resultados de caracterización de una población microbiana en el proceso de producción de etanol en una destilería colombiana, generan información útil en la búsqueda de estrategias para el control de la contaminación en el proceso de fermentación. Es necesario adelantar estudios que permitan la caracterización de agentes con efectos antimicrobianos, capaces de combatir, eficientemente, las bacterias contaminantes. Adicionalmente, se requiere la estandarización de técnicas, que permitan la identificación bacteriana con mayor precisión y en un menor tiempo.
AGRADECIMIENTOS
Agradecemos la generosa colaboración de la destilería colombiana que suministró las muestras y la información requerida para la elaboración del presente estudio. Además, al Comité de Investigación y Posgrados de la Facultad de Ciencias de la Universidad de Los Andes, por su apoyo financiero para la ejecución del mismo. Conflictos de intereses: El manuscrito fue preparado y revisado con la participación de todos los autores, quienes declaramos que no existe ningún conflicto de intereses que ponga en riesgo la validez de los resultados presentados.
BIBLIOGRAFÍA
1. ARIAS L., A.F.; ARBELÁEZ S., F.; ORTEGA A., J.D. 2006. Estrategia de desarrollo de biocombustibles: Implicaciones para el sector agropecuario. República de Colombia Ministerio de Agricultura y Desarrollo Rural. República de Colombia. p.9.
2. BAYROCK, D.; INGLEDEW, W. 2004. Inhibition of yeast by lactic acid bacteria in continuous culture: nutrient depletion and/or acid toxicity? J. Industrial Microbiol. & Biotechn. 31:362-368.
3. DENNIS, P.; INGLEDEW, W. 2005. Ethanol production in multistage continuous, single stage continuous, Lactobacillus-contaminated continuous, and batch fermentations. World J. Microbiol. & Biotechn. 21:83-88.
4. DUARTE, R.; BARROS, R.; FACKLAM, R.; TEIXEIRA, L. 2005. Phenotypic and Genotypic Characteristics of Streptococcus porcinus Isolated from Human Sources. J. Clinical Microbiol. 43(9):4592-4601.
5. DUBERNET, S.; DESMASURES, N.; GUÉGUEN, M. 2002. A PCR-based method for identification of lactobacilli at the genus level. FEMS Microbiol. Letters. 214:271-275.
6. GLAZER, A.; NIKAIDO H., 1998. Microbial Biotechnology: Fundamentals of Applied Microbiology. Ed. W.H. Freeman & Co. (USA). p.362-368.
7. GNECCO, J. 2006. Situación de la producción de alcohol en Colombia. Disponible desde Internet en: http://www.ciat.cgiar.org/training/pdf/060315_escenarios_de_produccion_de_etanol_en_colombia.pdf (con acceso 24/09/09).
8. HOLT, J.; KRIEG, N.; SNEATH, P.; STALEY, J.; WILLIAMS, S. 1994. Bergey's Manual of Determinative Bacteriology. Ed. Board (United States of America). p.565, 569.
9. HYNES, S.H.; KJARSGAARD, D.M.; THOMAS, K.C.; INGLEDEW, W.M. 1997. Use of virginiamycin to control the growth of lactic acidbacteria during alcohol fermentation. J. Industrial Microbiol. & Biotechn. 18:284-291.
10. KANDLER, O.; WEISS N. 1984. Regular, Nonsporing Gram-Positive Rods. En: Holt, J.; Sneath, P.; Mair, N.; Sharpe, E. eds. Bergey's Manual of Systematic Bacteriology. Ed. Board. (United States of America). p.1222-1230.
11. LARS, A. 2004. Lactic Acid Bacteria: Classification and Physiology. En: Salminen, S.; Wright, A.; Ouwehand, A. eds. Lactic Acid Bacteria Microbiological and functional aspects. Ed. Board (New York U.S.A.). p.4-8.
12. NARENDRANATH, N.; HYNES, S.; THOMAS, K.; INGLEDEW, W. 1997. Effects of Lactobacilli on yeast-catalyzed ethanol fermentations. Appl. and Environm. Microbiol. 63:4158-4163.
13. PRAKASH, O.; VERMA, M.; SHARMA, P.; KUMAR, M.; KUMARI, K.; SINGH, A.; KUMARI, H.; JIT, S.; GUPTA, S.; KHANNA, M.; LAL, R. 2007. Polyphasic approach of bacterial classifification - An overview of recent advances. Indian J. Microbiol. 47:98-108.
14. RIXON, J.; WARNER, P. 2003. Introduction: Background, revelant genetic techniques, and Terms. En: Wood, B.; Warner, P. eds. Genetics of lactic acid bacteria. New York. Chapter 1. p.3-15.
15. ROVIRA, J.; ROVIRA, A. 1994. Factores de inhibición del crecimiento de bacterias ácido lácticas. Alimentaria: Revista de Tecnología e Higiene de los alimentos. 254:31-36.
16. SINGH, S.; GOSWAMI, P.; SINGH, R.; HELLER, K. 2008. Application of molecular identification tools for Lactobacillus, with a focus on discrimination between closely related species: A review. LWT - Food Science and Technology. 42:448-457.
17. SKINNER, K.; LEATHERS, T. 2004. Bacterial contaminants of fuel ethanol production. J. Industrial Microbiol. Biotechnol. 31:401-408.
Recibido: Enero 31 de 2009; Aceptado: Octubre 13 de 2009

Revista U.D.C.A Actualidad & Divulgación Científica por Universidad de Ciencias Aplicadas y Ambientales se distribuye bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.