CIENCIAS DE LA SALUD-Artículo Científico
ANÁLISIS EVOLUTIVO DE LA ADHESIÓN: EVIDENCIA DE SELECCIÓN POSITIVA OPERANTE SOBRE EL LOCUS AlpAB Y EL GEN horB DE Helicobacter pylori
MOLECULAR EVOLUTIONARY ANALYSIS OF ADHERENCE: EVIDENCE OF POSITIVE SELECTION OPERATING ON AlpAB LOCUS AND horB ADHESINS OF Helicobacter pylori
Andrés Julián Gutiérrez-Escobar1
1Biólogo, Maestría en Ciencias Básicas Médicas. Docente Investigador Facultad de Medicina U.D.C.A, Coordinador Laboratorio de Biología Molecular, Grupo de Ciencias Básicas y Genética Humana Aplicada (GIBGA). Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, Bogotá, Colombia. E-mail: andresgutierrez@colombia.com
Rev. U.D.C.A Act. & Div. Cient. 16(1): 3 - 15, 2013
RESUMEN
La adherencia es un proceso clave para la infección por Helicobacter pylori, regulada por la expresión coordinada de adhesinas. Dentro de este grupo encontramos las proteínas AlpA/B y HorB que, recientemente, han llamado la atención de la comunidad científica, por su papel en el fitness de la bacteria. El objetivo del presente estudio fue describir la historia evolutiva de AlpA/B y HorB, desde un enfoque filogeográfico. Para este fin, se descargaron secuencias específicas para AlpA, AlpB y HorB desde Uniprot y fueron analizadas en Muscle, Mega 5.1, PhyML 3.0.1, DNAsp 5.0 y PAML 4.5. En este estudio, se identificó que los genes alpA, alpB y horB siguen un modelo evolutivo de nacimiento y de muerte de genes, bajo selección purificadora. Además, se detectaron patrones de diferenciación genética significativos para alpA, entre las poblaciones de América-Europa (FsT 0,51395) y de Europa-Asia (FsT 0,46030) y para alpB, entre las poblaciones de América-Europa (FsT 0,43711), datos soportados por la prueba McDonald y Kreitman. Seguidamente, mediante la herramienta PAML 4.5, se detectó que un 9,51% de la proteína AlpA, un 8,5% de la proteína AlpB y un 5% de la proteína HorBestán bajo selección positiva, determinando la presencia de linajes selectivos de H. pylori para zonas geográficas específicas, que favorecen la adherencia al hospedero. Finalmente, se concluye que los genes codificantes para las adhesinas AlpAB y HorB tienen procesos de adaptación locales correlacionados con los orígenes filogeográficos de las cepas analizadas.
Palabras clave: Adhesión, locus AlpAB, selección positiva, hipótesis de la reina roja, coevolución.
SUMMARY
The adherence to the epithelial gastric cell is a key step in the physiopathology of Helicobacter pylori infection. There are several outer membrane proteins involved in the adherence that are expressed in a sequential fashion, meanwhile the attachment to the host cell takes place. Currently, the adhesins AlpA, AlpB and HorB have attracted the attention because of i) its role in the adherence, ii) The differential activation of intracellular pathways in the gastric host epithelial cell between Asian and European strains and iii) in its role in the fitness according to gene deletion studies. It is largely known that H. pylori have a global distribution with regional divergent populations that have evolved following host conditions. Here, it was indentified that the evolutionary history of alpA, alpB and HorB adhesins followed a birth and death evolution model under purifying selection in which the deleterious mutations have been actively purged from the population allowing the fixation of beneficial changes for the fitness. It was detected based on permutation tests that H. pylori´s populations from America-Europe (FsT0.51395.) and Asia-Europe (FsT 0.46030) for alpA and the populations from America-Europe (FsT 0.43711) for alpB present a significant genetic differentiation mediated by positive selection as suggest by the MKT test results. Finally, it was found using PAML that a 9.51% of alpA, a 8.5% of alpB and a 5% of HorB proteins evolved under strong specific positive selection following linage specific geographic patterns.
Key words: Adhesion, AlpAB locus, molecular evolution, positive selection, Red Queen hypothesis, coevolution.
INTRODUCCIÓN
Helicobacter pylori es una bacteria Gram-negativa que hace parte de la microbiota normal del estómago humano (Bik et al. 2006), ya que no siempre desarrolla un proceso infeccioso (Mbulaiteye et al. 2009); sin embargo, cuando se presenta una infección activa, se asocia con úlcera, con gastritis y con cáncer gástrico (Blaser et al. 1995).
La mucosa gástrica es un nicho hostil y cambiante para la mayoría de patógenos bacterianos; no obstante, las bacterias del género Helicobacter han logrado adaptarse por medio de la regulación de la respuesta inflamatoria del hospedero (Algood & Cover, 2006; Kusters et al. 2006) y por sus características evolutivas.
Las poblaciones de H. pylori se caracterizan por ser genéticamente diversas, evidenciar ritmos de evolución rápidos, ser recombinantes, estar geográficamente aisladas siguiendo el patrón de las migraciones humanas y presentar modos de trasmisión intrafamiliares (Achtman et al. 1999; Falush et al. 2003; Kersulyte et al. 2000; Suerbaum et al. 1998). Estas características han ocasionado que las cepas evolucionen como linajes específicos en regiones geográficas particulares (Falush et al. 2003); es así, como se han identificado cepas típicas para África, Europa, Asia y América, con correlaciones clínicas diferenciales (Liu et al. 2005; Covacci et al. 1999).
La prevalencia de la infección y la especificidad regional de las cepas sugieren que H. pylori coevoluciona con su hospedero (Algood & Cover, 2006), por tanto, es factible plantear que los genes importantes para el éxito reproductivo de las poblaciones del patógeno evolucionan bajo selección positiva (Covacci et al. 1999). Ejemplos claros de este tipo de genes son los factores de virulencia cagA (Delgado et al. 2011), vacA, BabA, OpiA, SabA (Torres et al. 2010) y genes similares a Sel1 (Masako et al. 2007).
Algunos de los procesos bacterianos que mayor relación tienen con el éxito reproductivo (fitness) son la adherencia y la colonización a la célula epitelial hospedera. La adherencia de H. pylori a la célula epitelial gástrica es importante en la colonización del tejido, además, de participar en la respuesta inmune, tanto celular como humoral (Evans et al. 2000; Odenbreit, 2005).
De hecho, la expresión de factores proinflamatorios, como la IL-8, requiere de este contacto inicial, entre la bacteria y la célula blanco (Aihara et al. 1997). A la fecha, se han descrito un número de adhesinas, incluyendo BabA (Ilver et al. 1998), SabA (Mahdavi et al. 2002), OipA (Yamaoka et al. 2000), HopZ (Peck et al. 1999), AlpA/AlpB (Odenbreit et al. 1999; 2002) y HorB (Snelling et al. 2007).
Dentro del grupo de las adhesinas existen tres proteínas: AlpA, AlpB y HorB, cuya función ha sido caracterizada y estudiada en diferentes modelos experimentales. Las dos primeras conforman un operón y codifican para adhesinas, cuya actividad ha sido probada en cortes de tejido embebido en parafina, en cultivo celular, en modelo animal y en muestras de origen humano (Odenbreit et al. 1999). Recientemente, se ha identificado que la laminina, propia de la matriz extracelular del tejido gástrico, es el receptor para estas dos adhesinas (Senkovich et al. 2011).
Por otra parte, se ha demostrado el papel del locus AlpAB en la adhesión a la mucosa gástrica y en la expresión de interleucinas del hospedero (Odenbreit et al. 1999; Lu et al. 2007). En estos trabajos, se demostró que la deleción de estos genes reduce la expresión de IL-6 en células epiteliales gástricas y, además, que la reducción de la expresión de IL-8 es dependiente del origen filogeográfico de las cepas, observándose una diferencia significativa entre cepas del este asiático y cepas del oeste, lo que se conoce, actualmente, como la hipótesis este-oeste (Lu et al. 2007).
Las adhesinas AlpAB activan vías de señalización intracelular, mediada por quinasas, c-Fos y la proteína de respuesta al AMP cíclico; sin embargo, la activación del N-terminal de la quinasa Jun, c-jun y el NFkapaB fue exclusiva para las cepas del este asiático. Estos resultados sugieren que la variación geográfica de estas adhesinas se puede relacionar con las prevalencias observadas de cáncer gástrico, debido a los efectos diferenciales observados sobre la célula epitelial en cepas del este y del oeste, además de procesos evolutivos, a nivel molecular, que aún no han sido descritos para AlpAB (Kwong & Tiing, 2010). Finalmente, se ha determinado que HorB, además de ser una adhesina, es un factor de colonización importante para la bacteria (Snelling et al. 2007).
Derivado de los estudios anteriores, se sabe que la deleción de los genes alpA, alpB y HorB provoca una disminución significativa (entre el 60 al 70%) en la capacidad de adhesión del microorganismo a la célula hospedera, hecho que indica que estas enzimas son importantes para el éxito reproductivo del patógeno y quizás su ausencia podría limitar la supervivencia de la bacteria; no obstante, a la fecha, los procesos evolutivos que dan forma a los representantes modernos de estas tres proteínas para los diferentes linajes de H. pylori, se desconocen (Kwong & Tiing, 2010).
Es por esto que en el presente estudio, se investigó la dinámica evolutiva y poblacional de los genes alpA, alpB y HorB de H. pylori, a partir de cepas representantes de diferentes regiones geográficas. Se aplicaron modelos evolutivos de secuencias basados en codones en conjunción con modelos de máxima verosimilitud y se analizaron las relaciones entre polimorfismos específicos y sus implicaciones evolutivas. Se demostró que los tres genes presentan clara evidencia de una fuerte selección positiva operante, lo que indica que la capacidad de adhesión a la célula hospedera evoluciona de manera independiente y local para cada región geográfica.
MATERIALES Y MÉTODOS
En el presente trabajo, se siguió la metodología propuesta por Delgado et al. (2011), pero con algunas modificaciones leves. Brevemente, se realizó Blast (Altschul et al. 1997), en la base de datos Uniprot, con las siguientes secuencias semilla: O25570, para AlpA; O25595, para AlpB y O24941, para HorB de la cepa 26695, contra genomas representantes de diferentes poblaciones humanas, así: África J99 NC_000921; SouthAfrica7 NC_017361.1; Gambia 94/24 NC_017371.1; América: Cuz20 NC_017358.1; Shi470 NC_010698, V225d CP001582; Europa: 26695 NC_000915, HPGA1 NC_008086, P12 CP001217, G27 NC_011333; Asia: F16 NC_017368.1, 35A NC_017360.1, 51 NC_017382.1 e India7 NC_017372.1.
Las secuencias de los genomas, se eligieron según la epidemiología molecular, revisada en Suzuki et al. (2012). Las secuencias homólogas identificadas fueron alineadas con Muscle (Edgar, 2004), usando condiciones por defecto. Los alineamientos fueron inspeccionados visualmente y los formatos finales para los análisis evolutivos, se obtuvieron con Seaview (Gouy et al. 2010) y Jalview (Clamp et al. 2004).
Seguidamente, se usó Jmodeltest (Posada, 2008), para identificar el modelo evolutivo que mejor explicaba el alineamiento de ADN. Se usó el programa DNAsp5.0 (Librado & Rozas, 2009), para realizar las pruebas: MKT McDonald-Kreitman, Permutación (FsT bajo 50000 réplicas) y de recombinación. Finalmente, se aplicó MEGA 5.1 (Tamura et al. 2011), para la prueba Tajima (Tajima, 1989) y la prueba Z (Zhang et al. 1998).
Para la reconstrucción filogenética, se utilizó PhyML 3.0.1 (Guindon et al. 2010), estimando las opciones Ts/Tv, la proporción de sitios no variables y la distribución Gamma; la robustez estadística, se obtuvo mediante 1000 repeticiones Bootstrap. Los árboles filogenéticos se visualizaron en la herramienta Njplot (Perrière & Gouy, 1996); las filogenias también se desarrollaron por medio del método NeighborJoining (Saitou & Nei, 1987), bajo el modelo de distancia GTR, con 1000 repeticiones Bootstrap, aplicando MEGA 5.1 (Tamura et al. 2011).
Para el análisis de selección positiva los alineamientos fueron analizados usando el módulo codeml del programa PAML 4.5 (Yang, 2007). Se empleó el árbol descrito en la reconstrucción filogenética, además del generado por el servidor selecton (Stern et al. 2007), bajo el modelo M8a. Para el análisis de selección positiva, se emplearon las metodologías propuestas por Yang et al. 2005; 2000; Yang & Swanson, 2002; Yang, 1998; Yang & Nielsen, 2002; Zhang et al. 2005, utilizadas para el análisis de genes de H. pylori. Se emplearon los modelos evolutivos M0 (Neutral), M1 (casi neutral), M2 (selección), M3 (distribución discreta), M7 (distribución beta) y M8 (distribución beta, p y q). Se asumió un valor ω similar para cada rama del árbol, pero se permitió variación entre codones individuales de los genes, para medir la heterogeneidad de la variación de las presiones selectivas.
Se realizó la prueba Likelihood ratio test usando la herramienta Jmodel test. Se compararon los modelos M0/M1/M7 que no detectan sitios bajo la condición ω > 1 (selección positiva), con los modelos M2/M3/M8 que sí lo permiten; cada análisis fue repetido tres veces con diferentes valores iníciales ω, para evitar problemas de óptimos locales. Las comparaciones, se desarrollaron usando una distribución X2, bajo las indicaciones de Delgado et al. (2011) y las instrucciones de los desarrolladores de PAML 4.5. Todos los sitios con valores ω > 1 se consideraron bajo selección positiva. El mismo procedimiento, se llevó a cabo para cada subgrupo de genes correspondientes a regiones geográficas específicas.
RESULTADOS Y DISCUSIÓN
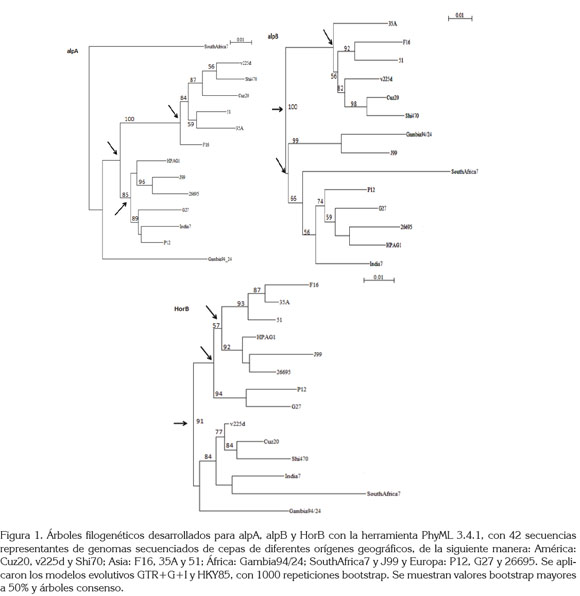
Análisis filogenético y poblacional de los genes alpA, alpB y HorB de H. pylori: La reconstrucción filogenética de los genes alpA, alpB y HorB replica, de manera relativamente fiel, la filogenia aceptada para H. pylori (Suzuki et al. 2012), logrando agrupar las cepas según origen geográfico, con valores bootstrap significativos. En los árboles, se observan múltiples eventos de duplicación (flechas) de genes, que dieron origen a los representantes modernos, con una mayor acumulación de cambios sinónimos que de cambios no sinónimos (alpApS = 0,081 y pN = 0,0031, alpBpS =0.1007 y pN = 0,0283, HorBpS= 0.0890 y pN= 0,0082), por lo que se puede sugerir que estas adhesinas siguen un modelo de evolución de nacimiento y muerte de genes, mediado por procesos de selección purificadora (Figura 1).

Los resultados de la prueba Tajima (D) son negativos para los tres genes, indicando una expansión poblacional con una baja frecuencia de polimorfismo, similar a lo que sucede después de eventos selectivos, como cuellos de botella y que es concordante con procesos de selección purificadora, lo cual, fue identificado con la prueba Z, que confirmó estos datos (Tabla 1).
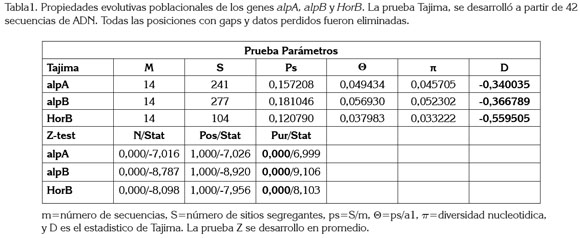
Bajo este mobdoeoltostreavpolmutaivyoo,resseae5li0m%inyanárbaolelelosscodnelseetnérseoo. s con efectos negativos para el "fitness" de las poblaciones del patógeno, lo que genera un espacio selectivo para la fijación de mutaciones benéficas. De esta manera, se podría sugerir que la capacidad de adherencia a la célula hospedera por parte de H. pylori, mediada por los genes alpA, alpB y HorB, está bajo un constante proceso de adaptación y de mejoramiento, en relación con su hospedero.
Recientemente, se identificaron marcadas diferencias funcionales entre cepas del este y del oeste de H. pylori con respecto a la activación de señales proinflamatorias en la célula hospedera, mediadas por el locus AlpAB (Lu et al. 2007), lo que sugiere la existencia de eventos selectivos variables entre cepas de diferentes regiones del globo. Los resultados de los análisis de permutación permitieron detectar diferencias significativas para alpA entre las poblaciones de América-Europa (FsT 0, 51395) y de Europa-Asia (FsT 0,46030); además, también se reporta, por primera vez, una diferencia significativa para el gen alpB, entre las poblaciones de América- Europa (FsT 0,43711) (Tabla 2A).
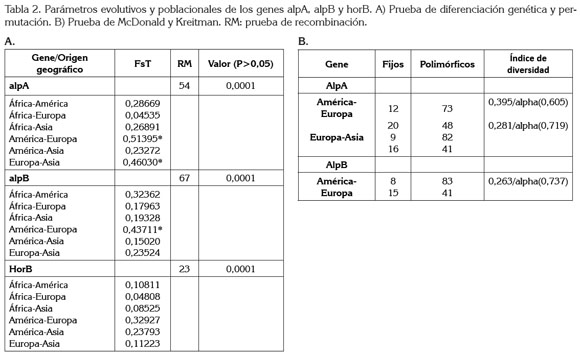
Con el análisis de los genes con la prueba de McDonald y Kreitman, se logró identificar, por primera vez, que las diferencias observadas por el test de permutación entre las poblaciones de América-Europa y de Europa-Asia para alpA y entre las poblaciones de América-Europa para alpB han ocurrido por eventos de selección direccional o selección positiva (Tabla 2B).
Eventos de selección positiva: Panorama mundial y por linajes regionales: Los modelos selectivos M2 y M3 lograron detectar sitios bajo selección positiva; sin embargo, su poder predictivo se afecta en casos de recombinación, proceso que es muy común en H. pylori y que ocurre en los tres genes estudiados en el presente estudio (Tabla 1). Por este motivo, se empleó el modelo M8, cuyo valor predictivo no se afecta por recombinación (Yang, 2007). Es importante aclarar que solamente se muestran los residuos bajo selección positiva identificados por el modelo, que mejor explica la evolución de las secuencias, después de aplicar la prueba LRT (Likeli- hood Radio Test) (Tabla 3).
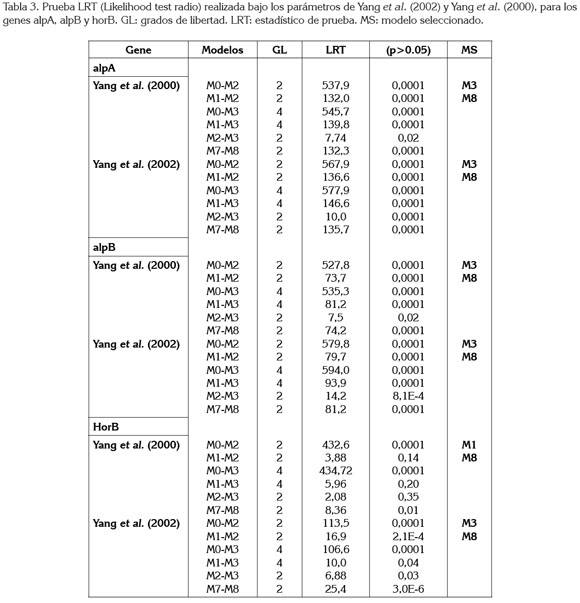
A nivel poblacional, los análisis realizados con la herramienta PAML indican que el 9,51% del gen alpA, el 8,5% del gen alpB y el 5% del gen están bajo selección positiva, lo cual, es significativo si se comparan con los estudios realizados para la región 3´ del gen cag con solamente un 6,95% o de la región central de babA, con un 9,4% (Masako et al. 2007); esto sugiere que un porcentaje significativo de las proteínas están siendo seleccionadas en las poblaciones estudiadas.
Hace poco tiempo, se determinó que la laminina es el ligando para las adhesinas AlpAB; sin embargo, esta proteína es de expresión constitutiva en el humano y, a diferencia de otras adhesinas, como BabA que tiene un receptor de expresión inducible, la interacción receptor ligando puede no ser la causa directa del tipo de selección natural (selección positiva), operante sobre el locus. Recientemente, se ha identificado que las proteínas AlpA, AlpB y HorB son inmunodominantes y quizás es uno de los factores que está produciendo la carrera adaptativa entre H. pylori y su hospedero.
Estos resultados son inquietantes, debido a que sugieren que estas tres adhesinas tienden a ser mega diversas a nivel poblacional y, además, están bajo constante selección positiva, lo que, probablemente, genere fenotipos adhesivos más virulentos y especializados, connotando un nuevo papel de la adhesión en la diferenciación local o regional de cepas (Tabla 4).
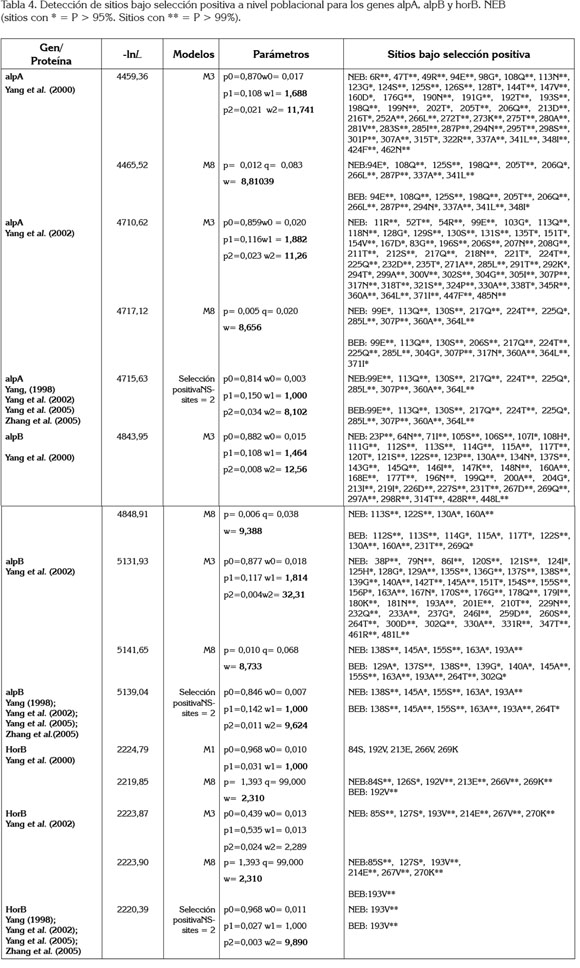
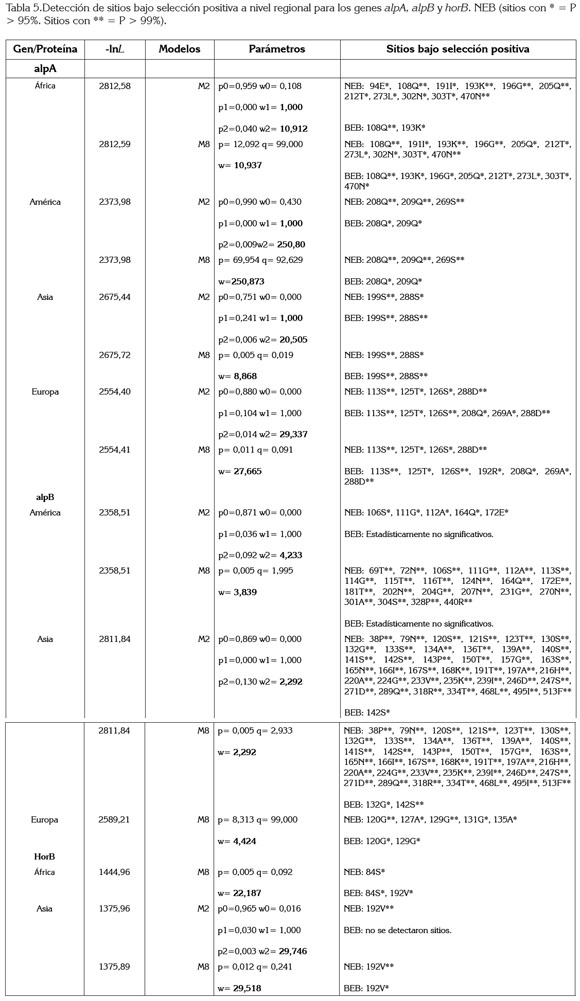
Seguidamente, los análisis de selección positiva realizados sobre las diferentes regiones geográficas del gen alpA, indican que cada una presenta su propia línea evolutiva con múltiples sitios bajo selección positiva, lo cual, sugiere y afianza los resultados arriba descritos de que los procesos selectivos son altamente locales y especializados para con esta enzima, constituyéndose quizás no solamente como un nuevo indicador de la virulencia, sino también, como un marcador diferenciador de cepas, a nivel local para las regiones.
Para el gen alpB, no se detectaron eventos selectivos para los representantes del linaje africano, pero sí se identificaron residuos bajo selección positiva para los linajes americano, asiático y europeo, respectivamente. Los eventos selectivos son particularmente intensos para el linaje asiático, mostrando múltiples sitios bajo selección positiva, lo que quizás se pueda correlacionar con las prevalencias de cáncer gástrico, halladas en la región. Finalmente, cabe mencionar que aunque se identificaron únicamente tres residuos bajo selección en los linajes africanos y asiáticos para el gen horB, éste es importante para el fitness de la especi, porque se constituye en un blanco terapéutico ideal (Tabla 5).
Síntesis evolutiva de la adhesión mediada por el locus AlpAB y HorB: El modo de evolución bajo selección purificadora purga fenotipos deletéreos en términos de la adhesión a la célula hospedera y, por ende, deja un nicho disponible para aquellos fenotipos con mejor capacidad de reconocimiento del hospedero. En el presente estudio, se sugiere que se pueden estar generando variantes locales del patógeno con patrones de adaptación regional, en términos de la adherencia, que son mediados por eventos de selección positiva sobre locus AlpAB y HorB, que benefician el fitness de las poblaciones del patógeno; estos cambios son diferencialmente locales, significando que las cepas de cada región geográfica evolucionan de manera independiente y, posiblemente, presentan un patrón de adhesión propio, efecto que necesita ser evaluado con mayor profundidad.
El locus AlpAB y el gen HorB están siendo perfeccionados al interactuar con factores del hospedero que, posiblemente, también están evolucionando bajo presiones evolutivas locales, estableciendo, de esta manera, una competencia o carrera coevolutiva entre patógeno y hospedero, ya que ambas especies presentan una velocidad diferencial adaptativa, en la que el patógeno persigue a su hospedero, que se podría explicar bajo la hipótesis coevolutiva de la reina roja (Woolhouse et al. 2002).
Agradecimientos: Agradezco al Doctor Juan Carlos Morales, al Doctor Carlos Barragán y al Doctor Martin Bayona, por sus valiosos aportes, tanto teóricos como de estilo, para la consecución del presente trabajo. Conflicto de intereses: El manuscrito fue preparado y revisado por el autor, quien declara que no existe ningún conflicto de interés que ponga en riesgo la validez de los resultados presentados. Financiación: Este estudio es parte de una investigación en curso aprobada y financiada por la Universidad de Ciencias Aplicadas y Ambientales U.D.C.A, en agosto de 2012.
BIBLIOGRAFÍA
1. ACHTMAN, M.; AZUMA, T.; BERG DE, ITO.; MORELLI, G.; PAN, Z.; SUERBAUM, S.; THOMPSON, S.; VAN DER ENDE, A.; VAN DOORN, L. 1999. Recombination and clonal groupings within Helicobacter pylori from different geographical regions. Mol. Microbiol. (Alemania). 32(3):459-470.
2. AIHARA, M.; TSUCHIMOTO, D.; TAKIZAWA, H.; AZUMA, A.; WAKEBE, H.; OHMOTO, Y.; IMAGAWA, K.; KIKUCHI, M.; MUKAIDA, N.; MATSUSHIMA, K. 1997. Mechanisms involved in Helicobacter pylori- induced interleukin-8 production by a gastric cancer cell line, MKN45. Infect.Immun. (Japan) 65(8):3218-3224.
3. ALGOOD, H.; COVER, T. 2006. Helicobacter pylori persistence: An overview of interactions between H. pylori and host immune defenses. Clin. Microbiol. Rev (USA).19(4):597-613.
4. ALTSCHUL, S.; MADDEN, T.; SCHÄFFER, A.; ZHANG, J.; ZHANG, Z.; MILLER, W.; LIPMAN, D. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. (USA). 25(17):3389-3402.
5. BIK, E.M.; ECKBURG, P.B.; GILL, S.R.; NELSON, K.E.; PURDOM, E.A.; FRITZ, F.; PEREZ-PEREZ, G.; BLASER, M.J.; RELMAN, D.A. 2006. Molecular analysis of the bacterial microbiota in the human stomach. Proc. Nal. Acad. Sci. 103(3):732-737.
6. BLASER, M.; PEREZ, G.; KLEANTHOUS, H.; COVER, T.; PEEK, R.; CHYOU, P.; STEMMERMANN, Y.; NOMURA, A. 1995. Infection with Helicobacter pylori strains processing cagA is associated with an increased risk of developing adenocarcinoma of the stomach. Cancer Res. (USA). 55(10):2111-2115.
7. CLAMP, M.; CUFF, J.; SEARLE, S.; BARTON, G. 2004. The Jalview Java Alignment Editor. Bioinformatics (Inglaterra). 20(3):426-437.
8. COVACCI, A.; TELFORD, J.L.; GIUDICE, G.D.; PARSONNET, J.; RAPPUOLI, R. 1999. Helicobacter pylori virulence and genetic geography. Science (Italia). 284(5418):1328-1333.
9. DELGADO-ROSADO, G.; DOMINGUEZ-BELLO, M.G.; MASSEY, S.E. 2011. Positive selection on a bacterial oncoprotein associated with gastric cancer. Gut Pathogen (Puerto Rico). 3:18-22.
10. EDGAR, R.C. 2004. Muscle: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32(5):1792-1797.
11. EVANS, D.J.; EVANS, D.G. 2000. Helicobacter pylori adhesins: review and perspectives. Helicobacter (USA). 5(4):183-195.
12. FALUSH, D.; WIRTH, T.; LINZ, B.; PRITCHARD, J.K.; STEPHENS, M.; KIDD, M.; BLASER, M.J.; GRAHAM, D.Y.; VACHER, S.; PEREZ-PEREZ, G.I.; YAMAOKA, Y.; MÉGRAUD, F.; OTTO, K.; REICHARD, U.; KATZOWITSCH, E.; WANG, X.; ACHTMAN, M.; SUERBAUM, S. 2003. Traces of human migrations in Helicobacter pylori populations. Science. (Alemania). 299(5612):1582-1585.
13. GOUY, M.; GUINDON, S.; GASCUEL, O. 2010. SeaView version 4: a multiplatform graphical user interface for sequence alignment and phylogenetic tree building. Mol.Biol.Evol. 27(2):221-224.
14. GUINDON, S.; DUFAYARD, J.F.; LEFORT, V.; ANISIMOVA, M.; HORDIJK, W.; GASCUEL, O. 2010. New Algorithms and Methods to Estimate MaximumLikelihood Phylogenies: Assessing the Performance of PhyML3.0. Syst. Biol. (Francia). 59(3):307-321.
15. ILVER, D.; ARNQVIST, A.; OGREN, J.; FRICK, I.M.; KERSULYTE, D.; INCECIK, E.T.; BERG, D.E.; COVACCI, A.; ENGSTRAND, L.; BORÉN, T. 1998. Helicobacter pylori adhesin binding fucosylated histo-blood group antigens revealed by retagging. Science. 279(5349):373-377.
16. KERSULYTE, D.; MUKHOPADHYAY, A.K.; VELAPATIÑO, B.; SU, W.; PAN, Z.; GARCIA, C.; HERNANDEZ, V.; VALDEZ, Y.; MISTRY, R.S.; GILMAN, R.H.; YUAN, Y.; GAO, H.; ALARCÓN, T.; LÓPEZ-BREA, M.; BALAKRISH NAIR, G.; CHOWDHURY, A.; DATTA, S.; SHIRAI, M.; NAKAZAWA, T.; ALLY, R.; SEGAL, I.; WONG, B.C.; LAM, S.K.; OLFAT, F.O.; BORÉN, T.; ENGSTRAND, L.; TORRES, O.; SCHNEIDER, R.; THOMAS, J.E.; CZINN, S.; BERG, D.E. 2000. Differences in genotypes of Helicobacter pylori from different human populations. J. Bacteriol. (USA). 182(11):3210-3218.
17. KUSTERS, J.G.; VAN VLIET, A.H.M.; KUIPERS, E.J. 2006. Pathogenesis of Helicobacter pylori infection. Clin. Microbiol. Rev (Holanda). 19(3):449-490.
18. KWONG MING, F.; TIING LEONG, A. 2010. Epidemiology of Helicobacter pylori infection and gastric cancer in Asia. J. Gastroent. Hepatol. (China). 25(3):479-486.
19. LIBRADO, P.; ROZAS, J. 2009. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data. Bioinformatics (España). 25(11):1451-1462.
20. LIU, Y.; PONSIOEN, C.I.J.; XIAO, S.; TYTGAT, G.N.J.; TEN KATE, F.J.W. 2005. Geographic pathology of Helicobacter pylori gastritis. Helicobacter. 10(2):107-113.
21. LU, H.; WU, J.Y.; BESWICK, E.J.; OHNO, T.; ODENBREIT, S.; HAAS, R.; REYES, V.E.; KITA, M.; GRAHAM, D.Y.; YAMAOKA, Y. 2007. Functional and intracellular signaling differences associated with the Helicobacter pylori AlpAB adhesin from Western and East Asian strains. J. Biol. Chem. (USA). 282(9):6242-6254.
22. MAHDAVI, J.; SONDÉN, B.; HURTIG, M.; OLFAT, F.O.; FORSBERG, L.; ROCHE, N.; ANGSTROM, J.; LARSSON, T.; TENEBERG, S.; KARLSSON, K.A.; ALTRAJA, S.; WADSTRÖM, T.; KERSULYTE, D.; BERG, D.E.; DUBOIS, A.; PETERSSON, C.; MAGNUSSON, K.E.; NORBERG, T.; LINDH, F.; LUNDSKOG, B.B.; ARNQVIST, A.; HAMMARSTRÖM, L.; BORÉN, T. 2002. Helicobacter pylori SabA adhesin in persistent infection and chronic inflammation. Science. (Suecia). 297(5581):573-578.
23. MASAKO OGURA, J.; PEREZ, C.; MITTL, P.R.E.; HAEKYUNG, L.E.E.; GEIDRIUS SHUMIN, T.; YOSHIYUKI, I.T.O.; OUSMAN, S.; DAIVA, D.; KALYANI, PUTTY.; BERG, D.; AWDHESH, K. 2007. Helicobacter pylori Evolution: Lineage-Specific Adaptations in Homologs of Eukaryotic Sel1-Like Genes. PLoSComput. Biol. (USA). 3(8):e151.
24. MBULAITEYE, S.M.; HISADA, M.; EL-OMAR, E.M. 2009. Helicobacter pylori associated global gastric cancer burden. Front. Biosci. (USA). 14:1490-1504.
25. ODENBREIT, S. 2005. Adherence properties of Helicobacter pylori: impact on pathogenesis and adaptation to the host. Int. J. Med. Microbiol. (Alemania). 295(5):317-324.
26. ODENBREIT, S.; FALLER, G.; HAAS, R. 2002. Role of the alpAB proteins and lipopolysaccharide in adhesion of Helicobacter pylori to human gastric tissue. Int. J. Med. Microbiol. 292(3-4):247-256.
27. ODENBREIT, S.; TILL, M.; HOFREUTER, D.; FALLER, G.; HAAS, R. 1999. Genetic and functional characterization of the alpAB gene locus essential for the adhesion of Helicobacterpylori to human gastric tissue. Mol. Microbiol. 31(5):1537-1548.
28. PECK, B.; ORTKAMP, M.; DIEHL, K.D.; HUNDT, E.; KNAPP, B. 1999. Conservation, localization and expression of HopZ, a protein involved in adhesion of Helicobacter pylori. Nucleic Acids Res. 27(16):3325-3333.
29. PERRIÈRE, G.; GOUY, M. 1996. WWW-Query: An online retrieval system for biological sequence banks. Biochimie. (Francia) 78(5):364-369.
30. POSADA, D. 2008. jModelTest: Phylogenetic Model Averaging. Mol. Biol. Evol. 25(7):1253-1256.
31. SAITOU, N.; NEI, M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4(4):406-425.
32. SENKOVICH, O.A.; YIN, J.; EKSHYYAN, V.; CONANT, C.; TRAYLOR, J.; ADEGBOYEGA, P.; MCGEE, D.J.; RHOADS, R.E.; SLEPENKOV, S.; TESTERMAN, T.L. 2011. Helicobacter pylori AlpA and AlpB bind host laminin and influence gastric inflammation in gerbils. Infect. Immun. (USA).79(8):3106-3116.
33. SNELLING, W.J.; MORAN, A.P.; RYAN, K.A.; SCULLY, P.; MCGOURTY, K.; COONEY, J.C.; ANNUK, H.; O'TOOLE, P.W. 2007. HorB (HP0127) is a gastric epithelial cell adhesin. Helicobacter. 12(3):200-209.
34. STERN, A.; DORON-FAIGENBOIM, A.; PEREZ, E.; MARTZ, E.; BACHARACH, E.; PUPKO, P. 2007. Selecton 2007: advanced models for detecting positive and purifying selection using a Bayesian inference approach. Nucleic Acids Res. (Israel). 35(Web Server issue): W506–W511.
35. SUERBAUM, S.; SMITH, J.M.; BAPUMIA, K.; MORELLI, G.; SMITH, N.H.; KUNSTMANN, E.; DYREK, I.; ACHTMAN, M. 1998. Free recombination within Helicobacter pylori. Proc. Nal. Acad. Sci. (Alemania). 95(21):12619-12624.
36. SUZUKI, R.; SHIOTA, S.; YAMAOKA, Y. 2012. Molecular epidemiology, population genetics, and pathogenic role of Helicobacter pylori. Infect. Genet. Evol. (Japan). 12(2):203-213.
37. TAJIMA, F. 1989. Statistical methods to test for nucleotide mutation hypothesis by DNA polymorphism. Genetics (Japan).123(3):585-595.
38. TAMURA, K.; PETERSON, D.; PETERSON, N.; STECHER, G.; NEI, M.; KUMAR, S. 2011. MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Mol. Biol. Evol. (Japan). 28(10):2731-2739.
39. TORRES, A.; GIONO, S.; CAMORLINGA, M.; CARLOS, F.; VARGAS, C.; TORRES, J. 2010. Evolution of bacterial genes: Evidences of positive Darwinian selection and fixation of base substitutions in virulence genes of Helicobacter pylori. Infect. Gen. Evol. 10(6):764-776.
40. WOOLHOUSE, M.E.; WEBSTER, J.P.; DOMINGO, E.; CHARLES WORTH, B.; LEVIN, B.R. 2002. Biological and biomedical implications of the co-evolution of pathogens and their hosts. Nat. Genet. (Belgica). 32(4):569-577.
41. YAMAOKA, Y.; KWON, D.H.; GRAHAM, D.Y. 2000. A M(r) 34,000 proinflammatory outer membrane protein (oipA) of Helicobacter pylori. Proc. Nal. Acad. Sci. (USA). 97(13):7533-7538.
42. YANG, Z. 1998. Likelihood ratio tests for detecting positive selection and application to primate lysozyme evolution. Mol. Biol. Evol. 15(5):568-573.
43. YANG, Z.; NIELSEN, R. 2002. Codon-substitution models for detecting molecular adaptation at individual sites along specific lineages. Mol. Biol. Evol. 19(6):908-917.
44. YANG, Z.; NIELSEN, R.; GOLDMAN, N.; PEDERSEN, A.M.K. 2000. Codon-substitution models for heterogeneous selection pressure at amino acid sites. Genetics. 155(1):431-449.
45. YANG, Z.; SWANSON, W.J. 2002. Codon-substitution models to detect adaptive evolution that account for heterogeneous selective pressures among site classes. Mol. Biol. Evol. 19(1):49-57.
46. YANG, Z.; WONG, W.; NIELSEN, R. 2005. Bayes empirical Bayes inference of amino acid sites under positive selection. Mol. Biol. Evol. 22(4):1107-1118.
47. YANG, Z. 2007. PAML 4: a program package for phylogenetic analysis by maximum likelihood. Mol. Biol. Evol. 13(5):555-566.
48. ZHANG, J.; NIELSEN, R.; YANG, Z. 2005. Evaluation of an improved branch-site likelihood method for detecting positive selection at the molecular level. Mol. Biol. Evol. (USA). 22(12):2472-2479.
49. ZHANG, J.; ROSENBERG, H.F.; NEI, M. 1998. Positive Darwinian selection after gene duplication in primate ribonuclease genes. Proc. Nal. Acad. Sci. 95 (7): 3708-3713.
Recibido: Septiembre 19 de 2012 Aceptado: Marzo 26 de 2013

Revista U.D.C.A Actualidad & Divulgación Científica por Universidad de Ciencias Aplicadas y Ambientales se distribuye bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.